Resultat
Figur 1
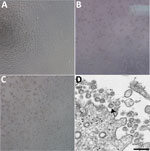
Figur 1. Cytopatisk effekt orsakad av coronavirus 2 med allvarligt akut respiratoriskt syndrom från patient med coronavirus-sjukdom, USA, 2020. A-C) Fas-kontrastmikroskopi av Vero-cellmonolager vid 3 dagar efter inokulering: A) Mock,…
En patient identifierades med bekräftad COVID-19 i delstaten Washington den 22 januari 2020. CPE observerades inte i mock-infekterade celler (figur 1, panel A). Cykeltröskelvärden (Ct) var 18-20 för NP-prover och 21-22 för OP-prover (1). De positiva kliniska proverna alikvoterades och återfrystes och inokulerades i cellkultur den 22 januari 2020. Vi observerade CPE 2 dagar efter inokulering och skördade viruslysat dag 3 efter inokulering (figur 1, panelerna B och C). Vi använde 50 μL av passage 1 viruslysat för extraktion av nukleinsyra för att bekräfta förekomsten av SARS-CoV-2 med hjälp av CDC:s molekylärdiagnostiska test (1). Ct-värdena för tre nukleinsyreextraktioner var 16,0-17,1 för nukleokapsiddel 1, 15,9-17,1 för nukleokapsiddel 2 och 16,2-17,3 för nukleokapsiddel 3, vilket bekräftade isoleringen av SARS-CoV-2 (Ct <40 anses vara ett positivt resultat). Vi testade också extrakt för ytterligare 33 olika luftvägspatogener med hjälp av Fast Track 33 Assay. Inga andra patogener upptäcktes. Identiteten stöddes dessutom av elektronmikroskopi i tunna sektioner (figur 1, panel D). Vi observerade en morfologi och morfogenes som är karakteristisk för coronavirus.
Vi använde isolat från den första passagen av ett OP- och ett NP-prov för helgenomsekvensering. Genomen från NP-exemplaret (GenBank accession MT020880) och OP-exemplaret (GenBank accession nr MT020881) visade 100 % identitet med varandra. Isolaten visade också 100 % identitet med motsvarande kliniska prov (GenBank accession no. MN985325).
Efter den andra passagen odlade vi inte OP- och NP-proverna separat. Vi passerade virusisolat ytterligare två gånger i Vero CCL-81-celler och titrerade genom att bestämma den 50 % vävnadskulturinfektiösa dosen (TCID50). Titrarna var 8,65 × 106 TCID50/mL för den tredje passagen och 7,65 × 106 TCID50/mL för den fjärde passagen.
Vi passerade detta virus i avsaknad av trypsin. Spikproteinsekvensen i SARS-CoV-2 har ett RRAR-instick vid S1-S2-gränssnittet som kan klyvas av furin (16). Högpatogena aviära influensavirus har högbasiska furin-klyvningsställen vid gränssnittet för hemagglutininprotein HA1-HA2 som möjliggör intracellulär mognad av virioner och effektivare viral replikation (17). RRAR-inslaget i SARS-CoV-2 kan ha en liknande funktion.
Figur 2
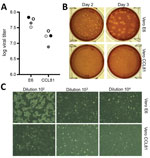
Figur 2. Viral spridning och kvantifiering av coronavirus 2 av allvarligt akut respiratoriskt syndrom från patient med coronavirus-sjukdom, USA, 2020. A) Två virusbestånd med passage 4 (svarta och grå cirklar) kvantifierades…
Vi genererade därefter ett bestånd med fjärde passage av SARS-CoV-2 på VeroE6-celler, en annan cellinje från en fetal rhesusapas njurar. Vi sekvenserade viralt RNA från SARS-CoV-2 passage 4 lager och bekräftade att det inte hade några nukleotidmutationer jämfört med den ursprungliga referenssekvensen (GenBank accession no. MN985325). SARS-CoV har visat sig växa bra på VeroE6-celler och MERS-CoV på Vero CCL81-celler (18,19). För att etablera en plackanalys och bestämma vilken Vero-celltyp som är att föredra för kvantifiering, titerade vi vårt passage 4-stock på VeroE6- och VeroCCL81-celler. Efter infektion med en utspädningsserie replikerades SARS-CoV-2 i båda Vero-celltyperna; virustitrarna var dock något högre i VeroE6-celler än i Vero CCL81-celler (figur 2, panel A). Dessutom var placken tydligare och mer synliga på Vero E6-cellerna (figur 2, panel B). Redan två dagar efter inokulering producerade VeroE6-cellerna distinkta plack som var synliga genom färgning med neutralt rött. Däremot producerade Vero CCL81-celler mindre tydliga plack och var lättast att kvantifiera genom färgning med neutralrött 3 dagar efter inokulering. På de enskilda plackmonolagerna producerade SARS-CoV-2-infektion av Vero E6-celler CPE med områden med cellutslag (figur 2, panel C). Däremot hade Vero CCL81-celler områden med döda celler som hade smält samman och bildat plack, men cellerna var inte klara. Tillsammans tyder dessa resultat på att VeroE6-celler kan vara det bästa valet för amplifiering och kvantifiering, men båda Vero-celltyperna stöder amplifiering och replikation av SARS-CoV-2.
Figur 3
Figur 3. Cellinjer från en patient med coronavirus-sjukdom, USA, 2020, mottagliga för SARS-coronavirus 2 (SARS-CoV-2). Cellinjerna infekterades med en hög infektionsmultiplicitet (>5), tvättades efter adsorption och…
Då forskning har inletts för att studera och reagera på SARS-CoV-2 behövs information om cellinjer och typer som är mottagliga för infektion. Därför undersökte vi SARS-CoV-2:s förmåga att infektera och replikera i flera vanliga primat- och humancellinjer, inklusive humana adenocarcinomceller (A549), humana leverceller (HUH7.0) och humana embryonala njurceller (HEK-293T), förutom Vero E6- och Vero CCL81-celler. Vi undersökte också en tillgänglig cellinje från stora bruna fladdermusens njurar (EFK3B) med avseende på replikationsförmåga för SARS-CoV-2. Varje cellinje inokulerades vid hög infektionsmultiplicitet och undersöktes 24 timmar efter infektionen (figur 3, panel A). Ingen CPE observerades i någon av cellinjerna utom i Vero-cellerna, som växte till >107 PFU 24 timmar efter infektionen. HUH7.0- och 293T-celler uppvisade däremot endast en blygsam virusreplikation, och A549-cellerna var inkompatibla med SARS-CoV-2-infektion. Dessa resultat stämmer överens med tidigare fynd av känslighet för SARS-CoV och tyder på att andra vanliga odlingssystem, inklusive MDCK, HeLa, HEP-2, MRC-5-celler och embryonerade ägg, sannolikt inte stöder replikation av SARS-CoV-2 (20-22). Dessutom replikerade SARS-CoV-2 inte i fladdermus EFK3B-celler, som är mottagliga för MERS-CoV. Tillsammans tyder resultaten på att SARS-CoV-2 upprätthåller en liknande profil som SARS-CoV när det gäller mottagliga cellinjer.
Efter att ha etablerat en robust infektion med SARS-CoV-2 i flera celltyper utvärderade vi härnäst korsreaktiviteten hos SARS-CoV-antikroppar mot SARS-CoV-2. Celllysat från infekterade cellinjer undersöktes för proteinanalys; vi fann att polyklonalt serum mot SARS-CoV spikprotein och nukleokapsidproteiner känner igen SARS-CoV-2 (figur 3, panelerna B och C). Nukleokapsidproteinet, som är mycket konserverat i hela grupp 2B-familjen, har >90% aminosyraidentitet mellan SARS-CoV och SARS-CoV-2. I överensstämmelse med replikationsresultaten (figur 3, panel A) visade SARS-CoV-2 ett robust nukleokapsidprotein i båda Vero-celltyperna, mindre protein i HUH7.0- och 293T-celler och minimalt protein i A549- och EFK3B-celler (figur 3, panel B). Antikroppen för SARS-CoV-spikprotein kände också igen SARS-CoV-2-spikprotein, vilket tyder på korsreaktivitet (figur 3, panel C). I överensstämmelse med SARS CoV observerades flera klyvda och oklyvda former av SARS-CoV-2 spikprotein. Klyvningsmönstret för den positiva SARS-spikekontrollen från Calu3-celler, en respiratorisk cellinje, varierar något och kan tyda på skillnader mellan proteolytisk klyvning av spikproteinerna mellan de två virusen på grund av ett förutspått införande av ett furin-klyvningsställe i SARS-CoV-2 (16). Skillnader i celltyp och förhållanden försvårar dock denna tolkning och visar på behovet av ytterligare studier i likvärdiga system. På det hela taget rekapitulerar proteinuttrycksdata från SARS-CoV nukleokapsid- och spikproteinantikroppar replikeringsfynden och indikerar att SARS-CoV-reagens kan användas för att karakterisera SARS-CoV-2-infektion.
Figur 4
Figur 4. Flerstegs tillväxtkurva för coronavirus 2 från patient med coronavirus-sjukdom, USA, 2020. Vero CCL81 (svart) och HUH7.0-celler (grönt) infekterades vid en multiplicitet av…
Slutligt utvärderade vi replikationskinetiken för SARS-CoV-2 i en tillväxtkurva i flera steg. I korthet infekterade vi Vero CCL-81- och HUH7.0-celler med SARS-CoV-2 vid en låg infektionsmultiplicitet (0,1) och utvärderade virusreplikationen var sjätte timme under 72 timmar efter inokulering, med separata skördar i de cellassocierade och supernatantkompartmenten (figur 4). I likhet med SARS-CoV replikerades SARS-CoV-2 snabbt i Vero-celler efter en inledande förmörkelsefas och uppnådde 105 TCID50/mL efter 24 timmars postinfektion och kulminerade vid >106 TCID50/mL. Vi observerade liknande titrar i cellassocierade och supernatantkompartment, vilket tyder på effektiv spridning. Trots högsta virala titrar vid 48 timmars postinokulering observerades inte större CPE förrän efter 60 timmars postinokulering och kulminerade vid 72 timmars postinokulering, vilket tyder på att infekterade monolager bör skördas innan högsta CPE observeras. Replikationen i HUH7.0-celler ökade också snabbt efter en inledande förmörkelsefas men nådde en platå efter 24 timmars postinokulering i den intracellulära avdelningen vid 2 × 103 TCID50/mL och minskade efter 66 timmars postinokulering. Viruset upptäcktes inte i supernatanten från infekterade HUH7-celler förrän efter 36 timmars postinokulering och uppvisade lägre titrar vid alla tidpunkter (figur 4). Större CPE observerades aldrig i HUH7.0-celler. Dessa resultat överensstämmer med tidigare rapporter om SARS-CoV och MERS-CoV, som tyder på liknande replikationsdynamik mellan de zoonotiska CoV-stammarna (23,24).
