Resultados
Figura 1
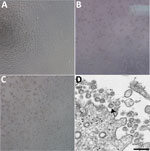
Figura 1. Efeito citopático causado pelo coronavírus 2 da síndrome respiratória aguda grave de paciente com doença coronavírus, Estados Unidos, 2020. A-C) Microscopia de contraste de fase de monocamadas de células Vero em 3 dias de pós-intinoculação: A) Mock,…
Um paciente foi identificado com COVID-19 confirmado no Estado de Washington a 22 de Janeiro de 2020. O CPE não foi observado em células infectadas com Mock (Figura 1, painel A). Os valores do limiar do ciclo (Ct) foram 18-20 para amostras NP e 21-22 para amostras OP (1). Os espécimes clínicos positivos foram aliquotados e recongelados inoculados em cultura celular em 22 de janeiro de 2020. Observamos CPE 2 dias pós-intinoculação e lisado viral colhido no dia 3 pós-intinoculação (Figura 1, painéis B, C). Utilizámos 50 μL de passagem 1 lisados virais para extracção de ácido nucleico para confirmar a presença do SRA-CoV-2 através do ensaio de diagnóstico molecular CDC (1). Os valores Ct de 3 extracções de ácido nucleico foram 16,0-17,1 para a porção nucleocápside 1, 15,9-17,1 para a porção nucleocápside 2, e 16,2-17,3 para a porção nucleocápside 3, o que confirmou o isolamento do SRA-CoV-2 (Ct <40 é considerado um resultado positivo). Também testamos extractos para 33 patogénios respiratórios diferentes adicionais usando o Ensaio Fast Track 33. Não foram detectados outros agentes patogénicos. A identificação foi adicionalmente apoiada por microscopia electrónica de secção fina (Figura 1, painel D). Observamos uma morfologia e morfogênese característica dos coronavírus.
Usamos isolados da primeira passagem de um OP e uma amostra NP para seqüenciamento do genoma inteiro. Os genomas da amostra NP (acesso GenBank MT020880) e da amostra OP (acesso GenBank no. MT020881) mostraram uma identidade de 100% um com o outro. Os isolados também mostraram 100% de identidade com a amostra clínica correspondente (GenBank accession no. MN985325).
Após a segunda passagem, não cultivamos amostras OP e NP separadamente. Passamos mais 2 vezes o isolamento do vírus em células Vero CCL-81 e titulamos através da determinação da dose infecciosa de 50% de cultura tecidual (TCID50). Os títulos foram 8,65 × 106 TCID50/mL para a terceira passagem e 7,65 × 106 TCID50/mL para a quarta passagem.
Passamos este vírus na ausência da tripsina. A sequência da proteína spike do SRA-CoV-2 tem uma inserção RRAR na interface S1-S2 que pode ser clivada pela furina (16). Os vírus da gripe aviária altamente patogénicos têm sítios de clivagem de furina altamente básicos na interface da proteína de hemaglutinina HA1-HA2 que permitem a maturação intracelular dos viriões e uma replicação viral mais eficiente (17). A inserção do RRAR na SRA-CoV-2 pode servir uma função semelhante.
Figure 2
Figure 2. Propagação viral e quantificação da síndrome respiratória aguda grave coronavírus 2 de paciente com doença coronavírus, Estados Unidos, 2020. A) Duas passagens de vírus 4 stocks (círculos negros e cinzentos) foram quantificados…
Gerámos subsequentemente uma quarta passagem de stock de células SRA-CoV-2 em células VeroE6, outra linha de células renais de macaco rhesus fetal. Sequenciámos o RNA viral da passagem 4 da SRA-CoV-2 e confirmámos que não havia mutações nucleotídicas em comparação com a sequência de referência original (acesso GenBank nº MN985325). Verificou-se que o SRA-CoV cresce bem em células VeroE6 e o MERS-CoV em células Vero CCL81 (18,19). Para estabelecer um ensaio de placa e determinar o tipo de célula Vero preferido para quantificação, titulamos o nosso stock de passagem 4 em células VeroE6 e VeroCCL81. Após infecção com uma série de diluição, o SRA-CoV-2 replicou-se em ambos os tipos de células Vero; contudo, os títulos virais foram ligeiramente mais elevados nas células VeroE6 do que nas células Vero CCL81 (Figura 2, painel A). Além disso, as placas eram mais distintas e visíveis nas células Vero E6 (Figura 2, painel B). Já em 2 dias de pós-intinoculação, as células VeroE6 produziram placas distintas visíveis através de coloração com vermelho neutro. Em contraste, as células Vero CCL81 produziram placas menos claras e foram mais facilmente quantificadas pela coloração com vermelho neutro 3 dias após atinoculação. Nas monocamadas individuais de placas, a infecção por SRA-CoV-2 das células Vero E6 produziu CPE com áreas de depuração celular (Figura 2, painel C). Em contraste, as células Vero CCL81 tinham áreas de células mortas que se tinham fundido para formar placas, mas as células não se depuravam. Em conjunto, estes resultados sugerem que as células VeroE6 podem ser a melhor escolha para amplificação e quantificação, mas ambos os tipos de células Vero suportam amplificação e replicação da SRA-CoV-2.
Figure 3
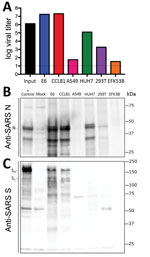
Figure 3. Linhas celulares de doentes com doença coronavírus, Estados Unidos, 2020, susceptíveis ao vírus corona da SRA 2 (SRA-CoV-2). As linhas celulares foram infectadas com uma elevada multiplicidade de infecção (>5), lavadas após adsorção, e…
Posto que a pesquisa foi iniciada para estudar e responder ao SRA-CoV-2, é necessária informação sobre as linhas celulares e tipos susceptíveis à infecção. Assim, examinámos a capacidade do SRA-CoV-2 de infectar e replicar em várias linhas de primatas e células humanas comuns, incluindo células adenocarcinoma humano (A549), células hepáticas humanas (HUH7.0) e células renais embrionárias humanas (HEK-293T), para além de células Vero E6 e Vero CCL81. Também examinámos uma linha de células renais de morcego pardo grande disponível (EFK3B) para a capacidade de replicação da SRA-CoV-2. Cada linha de células foi inoculada em alta multiplicidade de infecção e examinada 24 h após a infecção (Figura 3, painel A). Não foi observado CPE em nenhuma das linhas de células excepto nas células Vero, que cresceram para >107 PFU às 24 h pós-infecção. Em contraste, as células HUH7.0 e 293T apresentaram apenas uma replicação viral modesta, e as células A549 eram incompatíveis com a infecção pelo SRA-CoV-2. Estes resultados são consistentes com resultados de susceptibilidade anteriores para o SRA-CoV e sugerem que outros sistemas de cultura comuns, incluindo células MDCK, HeLa, HEP-2, MRC-5, e ovos embrionados, têm poucas probabilidades de suportar a replicação do SRA-CoV-2 (20-22). Adicionalmente, o SRA-CoV-2 não se replicou em células EFK3B do morcego, que são susceptíveis ao MERS-CoV. Em conjunto, os resultados indicam que o SRA-CoV-2 mantém um perfil semelhante ao do SRA-CoV em termos de linhas celulares susceptíveis.
Having established robust infection with SARS-CoV-2 in several cell types, we next evaluated the cross-reactivity of SARS-CoV antibodies against the SARS-CoV-2. Os lisados celulares das linhas celulares infectadas foram sondados para análise proteica; descobrimos que o soro policlonal contra a proteína do espigão do SRA-CoV e as proteínas da nucleocápside reconhecem o SRA-CoV-2 (Figura 3, painéis B, C). A proteína da nucleocápside, que está altamente conservada em todo o grupo 2B, retém >90% de identidade de aminoácidos entre o SRA-CoV e o SRA-CoV-2. Consistente com os resultados de replicação (Figura 3, painel A), o SRA-CoV-2 mostrou uma proteína nucleocápside robusta em ambos os tipos de células Vero, menos proteína em células HUH7.0 e 293T, e proteína mínima em células A549 e EFK3B (Figura 3, painel B). O anticorpo da proteína spike do SRA-CoV também reconheceu a proteína spike do SRA-CoV-2, indicando reactividade cruzada (Figura 3, painel C). Consistente com o CoV SRA, foram observadas várias formas clivadas e não clivadas da proteína do espigão do SRA-CoV-2. O padrão de clivagem do controlo positivo do espigão da SRA a partir das células de Calu3, uma linha de células respiratórias, varia ligeiramente e pode indicar diferenças entre as clivagens proteolíticas das proteínas do espigão entre os 2 vírus, devido a uma inserção prevista de um local de clivagem furina no SRA-CoV-2 (16). Contudo, as diferenças no tipo e condições celulares complicam esta interpretação e indicam a necessidade de mais estudos em sistemas equivalentes. Em geral, os dados de expressão proteica da nucleocapsida do SRA-CoV e dos anticorpos da proteína spike recapitulam os resultados da replicação e indicam que os reagentes do SRA-CoV podem ser usados para caracterizar a infecção pelo SRA-CoV-2.
Figure 4
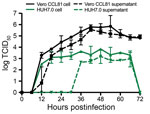
Figure 4. Curva de crescimento em várias etapas para o coronavírus 2 da síndrome respiratória aguda grave de um doente com doença coronavírus, Estados Unidos, 2020. As células Vero CCL81 (preto) e HUH7.0 (verde) foram infectadas numa multiplicidade de…
Finalmente, avaliamos a cinética de replicação do SRA-CoV-2 numa curva de crescimento em vários passos. Em resumo, infectamos células Vero CCL-81 e HUH7.0 com SRA-CoV-2 numa baixa multiplicidade de infecção (0,1) e avaliamos a replicação viral a cada 6 h para 72 h após atinoculação, com colheitas separadas nos compartimentos associados às células e sobrenadantes (Figura 4). Similar ao SRA-CoV, o SRA-CoV-2 replicou-se rapidamente em células Vero após uma fase inicial de eclipse, atingindo 105 TCID50/mL por 24 h após a infecção e atingindo um pico de >106 TCID50/mL. Observamos títulos semelhantes em compartimentos associados a células e sobrenadantes, que indicaram uma saída eficiente. Apesar do pico dos títulos virais por 48 h após atinoculação, o maior CPE não foi observado até 60 h após atinoculação e atingiu o pico de 72 h após atinoculação, indicando que as monocamadas infectadas devem ser colhidas antes do pico do CPE ser observado. A replicação em células HUH7.0 também aumentou rapidamente após uma fase inicial de eclipse, mas foi platugada por 24 h de pós-intinoculação no compartimento intracelular a 2 × 103 TCID50/mL e diminuiu após 66 h de pós-intinoculação. O vírus não foi detectado no sobrenadante de células HUH7 infectadas até 36 h pós-intinoculação e exibiu títulos mais baixos em todos os timepoints (Figura 4). O CPE principal nunca foi observado nas células do HUH7.0. Estes resultados são consistentes com relatórios anteriores da SRA-CoV e MERS-CoV, que sugeriram dinâmicas de replicação semelhantes entre as estirpes de CoV zoonótico (23,24).
