Results
Figure 1
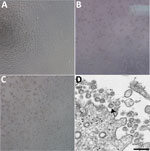
Figure 1. Effetto citopatico causato dal coronavirus 2 della sindrome respiratoria acuta grave da paziente con malattia da coronavirus, Stati Uniti, 2020. A-C) Microscopia a contrasto di fase di monostrati di cellule Vero a 3 giorni postinoculazione: A) Mock,…
Un paziente è stato identificato con COVID-19 confermato nello stato di Washington il 22 gennaio 2020. Il CPE non è stato osservato nelle cellule infettate mock (Figura 1, pannello A). I valori di soglia del ciclo (Ct) erano 18-20 per i campioni NP e 21-22 per i campioni OP (1). I campioni clinici positivi sono stati aliquotati e ricongelati inoculati in colture cellulari il 22 gennaio 2020. Abbiamo osservato il CPE 2 giorni dopo l’inoculazione e raccolto il lisato virale il giorno 3 dopo l’inoculazione (Figura 1, pannelli B, C). Abbiamo usato 50 μL di lisati virali di passaggio 1 per l’estrazione dell’acido nucleico per confermare la presenza di SARS-CoV-2 usando il test diagnostico molecolare CDC (1). I valori Ct di 3 estrazioni di acido nucleico erano 16,0-17,1 per la porzione di nucleocapside 1, 15,9-17,1 per la porzione di nucleocapside 2, e 16,2-17,3 per la porzione di nucleocapside 3, che hanno confermato l’isolamento di SARS-CoV-2 (Ct <40 è considerato un risultato positivo). Abbiamo anche testato gli estratti per altri 33 diversi patogeni respiratori utilizzando il Fast Track 33 Assay. Nessun altro patogeno è stato rilevato. L’identità è stata ulteriormente supportata dalla microscopia elettronica a sezione sottile (Figura 1, pannello D). Abbiamo osservato una morfologia e morfogenesi caratteristica dei coronavirus.
Abbiamo usato isolati dal primo passaggio di un OP e un campione NP per il sequenziamento del genoma intero. I genomi del campione NP (GenBank accession MT020880) e del campione OP (GenBank accession no. MT020881) hanno mostrato il 100% di identità tra loro. Gli isolati hanno anche mostrato il 100% di identità con il corrispondente campione clinico (GenBank accession no. MN985325).
Dopo il secondo passaggio, non abbiamo coltivato separatamente i campioni OP e NP. Abbiamo fatto passare l’isolato virale altre 2 volte in cellule Vero CCL-81 e abbiamo titolato determinando la dose infettiva del 50% della cultura dei tessuti (TCID50). I titoli erano 8,65 × 106 TCID50/mL per il terzo passaggio e 7,65 × 106 TCID50/mL per il quarto passaggio.
Abbiamo fatto passare questo virus in assenza di tripsina. La sequenza della proteina spike di SARS-CoV-2 ha un’inserzione RRAR all’interfaccia S1-S2 che potrebbe essere scissa dalla furina (16). I virus dell’influenza aviaria altamente patogeni hanno siti di scissione della furina altamente basici all’interfaccia della proteina emagglutinina HA1-HA2 che permettono la maturazione intracellulare dei virioni e una replicazione virale più efficiente (17). L’inserzione RRAR nella SARS-CoV-2 potrebbe avere una funzione simile.
Figura 2
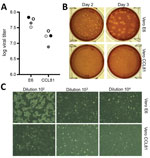
Figura 2. Propagazione virale e quantificazione del coronavirus 2 della sindrome respiratoria acuta grave da paziente con malattia da coronavirus, Stati Uniti, 2020. A) Due stock di passaggio 4 del virus (cerchi neri e grigi) sono stati quantificati…
In seguito abbiamo generato uno stock di quarto passaggio di SARS-CoV-2 su cellule VeroE6, un’altra linea cellulare fetale di rene di scimmia rhesus. Abbiamo sequenziato l’RNA virale dello stock di SARS-CoV-2 di quarto passaggio e abbiamo confermato che non presenta mutazioni nucleotidiche rispetto alla sequenza di riferimento originale (GenBank accession no. MN985325). È stato riscontrato che SARS-CoV cresce bene su cellule VeroE6 e MERS-CoV su cellule Vero CCL81 (18,19). Per stabilire un test della placca e determinare il tipo di cellula Vero preferito per la quantificazione, abbiamo titolato il nostro stock di passaggio 4 su cellule VeroE6 e VeroCCL81. Dopo l’infezione con una serie di diluizioni, la SARS-CoV-2 si è replicata in entrambi i tipi di cellule Vero; tuttavia, i titoli virali erano leggermente più alti nelle cellule VeroE6 che nelle cellule Vero CCL81 (Figura 2, pannello A). Inoltre, le placche erano più distinte e visibili sulle cellule Vero E6 (Figura 2, pannello B). Già 2 giorni dopo l’inoculazione, le cellule VeroE6 hanno prodotto placche distinte visibili mediante colorazione con rosso neutro. Al contrario, Vero CCL81 cellule prodotte meno chiare placche ed è stato più facilmente quantificato dalla colorazione con rosso neutro 3 giorni postinoculazione. Sui singoli monostrati di placche, l’infezione da SARS-CoV-2 delle cellule Vero E6 ha prodotto CPE con aree di clearance cellulare (Figura 2, pannello C). Al contrario, le cellule Vero CCL81 avevano aree di cellule morte che si erano fuse per formare placche, ma le cellule non si sono liberate. Insieme, questi risultati suggeriscono che le cellule VeroE6 potrebbero essere la scelta migliore per l’amplificazione e la quantificazione, ma entrambi i tipi di cellule Vero supportano l’amplificazione e la replicazione del SARS-CoV-2.
Figura 3
Figura 3. Linee cellulari da paziente con malattia da coronavirus, Stati Uniti, 2020, suscettibili al coronavirus 2 della SARS (SARS-CoV-2). Le linee cellulari sono state infettate con un’alta molteplicità di infezione (>5), lavate dopo l’adsorbimento, e…
Perché la ricerca è stata avviata per studiare e rispondere alla SARS-CoV-2, sono necessarie informazioni sulle linee cellulari e sui tipi suscettibili di infezione. Pertanto, abbiamo esaminato la capacità della SARS-CoV-2 di infettare e replicare in diverse linee cellulari comuni di primati e umane, tra cui cellule di adenocarcinoma umano (A549), cellule epatiche umane (HUH7.0) e cellule di rene embrionale umano (HEK-293T), oltre alle cellule Vero E6 e Vero CCL81. Abbiamo anche esaminato una linea cellulare di rene di pipistrello marrone grande (EFK3B) per la capacità di replicazione del SARS-CoV-2. Ogni linea cellulare è stata inoculata ad alta molteplicità di infezione ed esaminata 24 ore dopo l’infezione (Figura 3, pannello A). Non è stato osservato alcun CPE in nessuna delle linee cellulari tranne che nelle cellule Vero, che sono cresciute fino a >107 PFU a 24 ore dall’infezione. Al contrario, le cellule HUH7.0 e 293T hanno mostrato solo una modesta replicazione virale, e le cellule A549 erano incompatibili con l’infezione da SARS-CoV-2. Questi risultati sono coerenti con i precedenti risultati di suscettibilità per la SARS-CoV e suggeriscono che altri sistemi di coltura comuni, tra cui MDCK, HeLa, HEP-2, cellule MRC-5 e uova embrionate, non sono in grado di sostenere la replicazione della SARS-CoV-2 (20-22). Inoltre, la SARS-CoV-2 non si è replicata nelle cellule EFK3B di pipistrello, che sono suscettibili al MERS-CoV. Insieme, i risultati indicano che SARS-CoV-2 mantiene un profilo simile a quello di SARS-CoV in termini di linee cellulari suscettibili.
Avendo stabilito una robusta infezione con SARS-CoV-2 in diversi tipi di cellule, abbiamo poi valutato la cross-reattività degli anticorpi contro SARS-CoV-2. I lisati cellulari delle linee cellulari infettate sono stati analizzati per l’analisi delle proteine; abbiamo scoperto che il siero policlonale contro la proteina spike del SARS-CoV e le proteine del nucleocapside riconoscono il SARS-CoV-2 (Figura 3, pannelli B, C). La proteina del nucleocapside, che è altamente conservata in tutta la famiglia del gruppo 2B, conserva >90% di identità aminoacidica tra SARS-CoV e SARS-CoV-2. Coerentemente con i risultati della replicazione (Figura 3, pannello A), SARS-CoV-2 ha mostrato una proteina del nucleocapside robusta in entrambi i tipi di cellule Vero, meno proteina nelle cellule HUH7.0 e 293T, e una proteina minima nelle cellule A549 e EFK3B (Figura 3, pannello B). L’anticorpo della proteina spike del SARS-CoV ha riconosciuto anche la proteina spike del SARS-CoV-2, indicando una reattività incrociata (Figura 3, pannello C). Coerentemente con il SARS CoV, sono state osservate diverse forme scisse e non scisse della proteina di punta del SARS-CoV-2. Il modello di scissione del controllo positivo del picco di SARS proveniente dalle cellule Calu3, una linea cellulare respiratoria, varia leggermente e potrebbe indicare differenze tra la scissione proteolitica delle proteine del picco tra i 2 virus a causa dell’inserimento previsto di un sito di scissione della furina nel SARS-CoV-2 (16). Tuttavia, le differenze nel tipo di cellule e nelle condizioni complicano questa interpretazione e indicano la necessità di ulteriori studi in sistemi equivalenti. Nel complesso, i dati di espressione proteica degli anticorpi del nucleocapside e della proteina spike di SARS-CoV ricapitolano i risultati della replicazione e indicano che i reagenti di SARS-CoV possono essere usati per caratterizzare l’infezione da SARS-CoV-2.
Figura 4
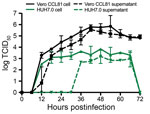
Figura 4. Curva di crescita multistep per il coronavirus 2 della sindrome respiratoria acuta grave da paziente con malattia da coronavirus, Stati Uniti, 2020. Le cellule Vero CCL81 (nero) e HUH7.0 (verde) sono state infettate a una molteplicità di…
Infine, abbiamo valutato la cinetica di replicazione del SARS-CoV-2 in una curva di crescita multistep. In breve, abbiamo infettato le cellule Vero CCL-81 e HUH7.0 con SARS-CoV-2 a una bassa molteplicità di infezione (0,1) e abbiamo valutato la replicazione virale ogni 6 ore per 72 ore dopo l’inoculazione, con raccolte separate nei compartimenti associati alle cellule e nel surnatante (Figura 4). Simile al SARS-CoV, il SARS-CoV-2 si è replicato rapidamente nelle cellule Vero dopo una fase iniziale di eclissi, raggiungendo 105 TCID50/mL entro 24 ore dall’infezione e raggiungendo un picco di >106 TCID50/mL. Abbiamo osservato titoli simili nei compartimenti associati alle cellule e nel surnatante, il che ha indicato un’efficiente uscita. Nonostante i titoli virali di picco entro 48 h postinoculazione, il CPE maggiore non è stato osservato fino a 60 h postinoculazione e ha raggiunto il picco a 72 h postinoculazione, indicando che i monostrati infetti dovrebbero essere raccolti prima di osservare il CPE di picco. La replicazione nelle cellule HUH7.0 è anche aumentata rapidamente dopo una fase iniziale di eclissi, ma si è stabilizzata entro 24 h postinoculazione nel compartimento intracellulare a 2 × 103 TCID50/mL ed è diminuita dopo 66 h postinoculazione. Il virus non è stato rilevato nel surnatante delle cellule HUH7 infette fino a 36 h postinoculazione e ha mostrato titoli più bassi in tutti i timepoint (Figura 4). Il CPE maggiore non è mai stato osservato nelle cellule HUH7.0. Questi risultati sono coerenti con i rapporti precedenti per SARS-CoV e MERS-CoV, che hanno suggerito dinamiche di replicazione simili tra i ceppi di CoV zoonotici (23,24).
