Tulokset
Kuva 1
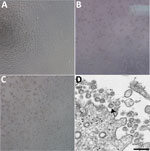
Kuva 1. Vakavan akuutin hengitystieoireyhtymän koronavirus 2:n aiheuttama sytopaattinen vaikutus potilaalta, jolla oli koronavirustauti, Yhdysvallat, 2020. A-C) Vaihekontrastimikroskopia Vero-solujen monolayereistä 3 päivää inokulaation jälkeen: A) Mock,…
Potilaalla todettiin vahvistettu COVID-19 Washingtonin osavaltiossa 22. tammikuuta 2020. CPE:tä ei havaittu mock-infektoiduissa soluissa (kuva 1, paneeli A). Syklikynnysarvot (Ct) olivat 18-20 NP-näytteissä ja 21-22 OP-näytteissä (1). Positiiviset kliiniset näytteet aliquotoitiin ja inokuloitiin uudelleen pakastettuna soluviljelmiin 22. tammikuuta 2020. Havaitsimme CPE:n 2 päivää inokulaation jälkeen ja keräsimme viruslysaatin 3. päivänä inokulaation jälkeen (kuva 1, paneelit B, C). Käytimme 50 μl läpiviennin 1 viruslysaattia nukleiinihapon uuttamiseen SARS-CoV-2:n esiintymisen varmistamiseksi CDC:n molekyylidiagnostisella määrityksellä (1). Kolmen nukleiinihappouuton Ct-arvot olivat 16,0-17,1 nukleokapsidin osalle 1, 15,9-17,1 nukleokapsidin osalle 2 ja 16,2-17,3 nukleokapsidin osalle 3, mikä vahvisti SARS-CoV-2:n eristämisen (Ct <40 pidetään positiivisena tuloksena). Testasimme uutteita myös 33 muun eri hengitystiepatogeenin varalta Fast Track 33 Assay -testillä. Muita taudinaiheuttajia ei havaittu. Identiteettiä vahvisti lisäksi ohutleikkauselektronimikroskopia (kuva 1, paneeli D). Havaitsimme koronaviruksille tyypillisen morfologian ja morfogeneesin.
Käytimme koko genomin sekvensointia varten isolaatteja OP- ja NP-näytteen ensimmäisestä läpikäynnistä. NP-näytteen (GenBank-tunniste MT020880) ja OP-näytteen (GenBank-tunniste MT020881) genomit olivat 100-prosenttisesti identtisiä keskenään. Isolaatit osoittivat myös 100-prosenttista identtisyyttä vastaavan kliinisen näytteen (GenBank-tunnistenumero MN985325) kanssa.
Toisen läpikäynnin jälkeen emme viljelleet OP- ja NP-näytteitä erikseen. Passagoimme virusisolaatin vielä 2 kertaa Vero CCL-81 -soluissa ja titrasimme määrittämällä 50 %:n kudosviljelyssä infektiivisen annoksen (TCID50). Titterit olivat 8,65 × 106 TCID50/ml kolmannessa passiossa ja 7,65 × 106 TCID50/ml neljännessä passiossa.
Passiivoimme tämän viruksen ilman trypsiiniä. SARS-CoV-2:n piikkiproteiinisekvenssissä on RRAR-insertio S1-S2-rajapinnassa, jonka furiini saattaa pilkkoa (16). Erittäin patogeenisilla lintuinfluenssaviruksilla on hemagglutiniiniproteiinin HA1-HA2-rajapinnassa erittäin emäksisiä furiinin pilkkoutumiskohtia, jotka mahdollistavat virionien solunsisäisen kypsymisen ja tehokkaamman virusreplikaation (17). SARS-CoV-2:n RRAR-insertiolla saattaa olla samanlainen tehtävä.
Kuva 2
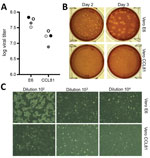
Kuva 2. SARS-CoV-2:n RRAR-insertio. Vakavan akuutin hengitystieoireyhtymän koronavirus 2:n viruksen leviäminen ja kvantitatiivinen määritys koronavirusta sairastavasta potilaasta, Yhdysvallat, 2020. A) Kaksi viruksen neljännen passage-kantaa (mustat ja harmaat ympyrät) kvantifioitiin…
Tuotimme sittemmin SARS-CoV-2:n neljännen passage-kannan VeroE6-soluilla, joka on toinen sikiöaikainen rhesusapinan munuaissolulinja. Sekvensoimme SARS-CoV-2:n neljännen läpiviennin viruksen RNA:n ja vahvistimme, että siinä ei ollut nukleotidimutaatioita verrattuna alkuperäiseen referenssisekvenssiin (GenBank-tunniste MN985325). SARS-CoV:n on todettu kasvavan hyvin VeroE6-soluissa ja MERS-CoV:n Vero CCL81-soluissa (18,19). Laatiaksemme plakkimäärityksen ja määrittääksemme, mikä Vero-solutyyppi on suositeltavin kvantifiointia varten, titteroimme läpivienti 4 -kantaamme VeroE6- ja VeroCCL81-soluilla. Laimennossarjan infektion jälkeen SARS-CoV-2 replikoitui molemmissa Vero-solutyypeissä; virustitterit olivat kuitenkin hieman korkeammat VeroE6-soluissa kuin VeroCCL81-soluissa (kuva 2, paneeli A). Lisäksi plakit olivat selvempiä ja näkyvämpiä Vero E6 -soluissa (kuva 2, paneeli B). Jo 2 päivää inokulaation jälkeen VeroE6-solut tuottivat selviä plakkeja, jotka näkyivät neutraalipunaisella värjäyksellä. Sitä vastoin Vero CCL81 -solut tuottivat vähemmän selkeitä plakkeja, ja ne oli helpointa määrittää värjäämällä neutraalipunalla 3 päivää inokulaation jälkeen. Vero E6 -solujen SARS-CoV-2-infektio tuotti yksittäisissä plakkimonolyyreissä CPE:tä, jossa oli solujen poistuma-alueita (kuva 2, paneeli C). Sitä vastoin Vero CCL81 -soluissa oli kuolleiden solujen alueita, jotka olivat sulautuneet plakkien muodostamiseksi, mutta solut eivät puhdistuneet. Yhdessä nämä tulokset viittaavat siihen, että VeroE6-solut saattaisivat olla paras valinta monistamiseen ja kvantifiointiin, mutta molemmat Vero-solutyypit tukevat SARS-CoV-2:n monistamista ja monistumista.
Kuva 3
Kuva 3. SARS-CoV-2:n monistaminen ja monistuminen. SARS-koronavirustautia sairastavan potilaan solulinjat, Yhdysvallat, 2020, jotka ovat alttiita SARS-koronavirukselle 2 (SARS-CoV-2). Solulinjat infektoitiin korkealla infektiokertoimella (>5), pestiin adsorption jälkeen ja…
Koska SARS-CoV-2:n tutkiminen ja siihen reagoiminen on aloitettu, tarvitaan tietoa infektiolle alttiista solulinjoista ja -tyypeistä. Siksi tutkimme SARS-CoV-2:n kykyä infektoida ja lisääntyä useissa tavallisissa kädellisten ja ihmisen solulinjoissa, kuten ihmisen adenokarsinoomasoluissa (A549), ihmisen maksasoluissa (HUH7.0) ja ihmisen alkion munuaissoluissa (HEK-293T) sekä Vero E6- ja Vero CCL81 -soluissa. Tutkimme myös saatavilla olevan ison ruskean lepakon munuaissolulinjan (EFK3B) SARS-CoV-2:n replikaatiokapasiteettia. Kukin solulinja inokuloitiin korkealla infektiokertoimella ja tutkittiin 24 tuntia infektion jälkeen (kuva 3, paneeli A). Yhdessäkään solulinjassa ei havaittu CPE:tä lukuun ottamatta Vero-soluja, jotka kasvoivat >107 PFU:hun 24 tuntia infektion jälkeen. Sitä vastoin HUH7.0- ja 293T-soluissa viruksen replikaatio oli vain vaatimatonta, ja A549-solut olivat yhteensopimattomia SARS-CoV-2-infektion kanssa. Nämä tulokset ovat yhdenmukaisia aiempien SARS-CoV:n herkkyyslöydösten kanssa ja viittaavat siihen, että muut yleiset viljelyjärjestelmät, kuten MDCK-, HeLa-, HEP-2-, MRC-5-solut ja alkiomunat, eivät todennäköisesti tue SARS-CoV-2:n replikaatiota (20-22). SARS-CoV-2 ei myöskään replikoitunut lepakon EFK3B-soluissa, jotka ovat herkkiä MERS-CoV:lle. Yhdessä nämä tulokset osoittavat, että SARS-CoV-2:lla on samanlainen profiili kuin SARS-CoV:llä alttiiden solulinjojen suhteen.
Kun olimme todenneet SARS-CoV-2:n vankan infektion useissa solutyypeissä, arvioimme seuraavaksi SARS-CoV:n vasta-aineiden ristireaktiivisuutta SARS-CoV-2:ta vastaan. Infektoituneista solulinjoista saadut solulysaatit tutkittiin proteiinianalyysiä varten; havaitsimme, että SARS-CoV:n piikkiproteiinia ja nukleokapsidiproteiineja vastaan suunnatut polyklonaaliset seerumit tunnistavat SARS-CoV-2:n (kuva 3, paneelit B, C). Nukleokapsidiproteiini, joka on erittäin konservoitunut koko ryhmän 2B-perheessä, säilyttää >90 prosentin aminohappoidentiteetin SARS-CoV:n ja SARS-CoV-2:n välillä. Replikaatiotulosten (kuva 3, paneeli A) mukaisesti SARS-CoV-2 osoitti vahvaa nukleokapsidiproteiinia molemmissa Vero-solutyypeissä, vähemmän proteiinia HUH7.0- ja 293T-soluissa ja minimaalista proteiinia A549- ja EFK3B-soluissa (kuva 3, paneeli B). SARS-CoV-piikkiproteiinivasta-aine tunnisti myös SARS-CoV-2:n piikkiproteiinin, mikä viittaa ristireaktiivisuuteen (kuva 3, paneeli C). SARS-CoV-2:n piikkiproteiinista havaittiin useita pilkkoutuneita ja pilkkoutumattomia muotoja SARS-CoV-2:n piikkiproteiinin tavoin. Hengitysteiden solulinjan Calu3-soluista saadun SARS-piikkiproteiinin positiivisen kontrollin pilkkoutumismalli vaihtelee hieman ja saattaa viitata eroihin näiden kahden viruksen piikkiproteiinien proteolyyttisessä pilkkoutumisessa, koska SARS-CoV-2:ssa on ennustettu furiinin pilkkoutumiskohdan lisäys (16). Solutyyppi- ja olosuhde-erot vaikeuttavat kuitenkin tätä tulkintaa ja osoittavat, että lisätutkimuksia on tehtävä vastaavissa järjestelmissä. Kaiken kaikkiaan SARS-CoV:n nukleokapsidi- ja piikkiproteiinivasta-aineiden proteiiniekspressiotiedot toistavat replikaatiohavainnot ja osoittavat, että SARS-CoV-reagensseja voidaan käyttää SARS-CoV-2-infektion karakterisointiin.
Kuva 4
Kuva 4. SARS-CoV-reagenssit. Vakavan akuutin hengitystieoireyhtymän koronavirus 2:n monivaiheinen kasvukäyrä koronavirusta sairastavasta potilaasta, Yhdysvallat, 2020. Vero CCL81 (musta) ja HUH7.0-solut (vihreä) infektoitiin moninkertaisuudella…
Viimeiseksi arvioimme SARS-CoV-2:n replikaatiokinetiikkaa monivaiheisella kasvukäyrällä. Lyhyesti sanottuna infektoimme Vero CCL-81- ja HUH7.0-solut SARS-CoV-2:lla matalalla infektiokerrannaisuudella (0,1) ja arvioimme viruksen replikaatiota 6 tunnin välein 72 tunnin ajan inokulaation jälkeen siten, että soluassosiaatio- ja supernatanttiosastoista tehtiin erilliset sadot (kuva 4). SARS-CoV:n tavoin SARS-CoV-2 replikoitui nopeasti Vero-soluissa alkuvaiheen pimennysvaiheen jälkeen saavuttaen 105 TCID50/ml 24 tuntia infektion jälkeen ja saavuttaen huippunsa >106 TCID50/ml. Havaitsimme samanlaisia tittereitä soluun assosioituneissa ja supernatanttiosastoissa, mikä viittaa tehokkaaseen egressioon. Vaikka virustitterit olivat huipussaan 48 tuntia inokulaation jälkeen, merkittävää CPE:tä havaittiin vasta 60 tuntia inokulaation jälkeen, ja se saavutti huippunsa 72 tuntia inokulaation jälkeen, mikä osoittaa, että infektoituneet monolayerit olisi kerättävä ennen kuin CPE:n huippu havaitaan. Replikaatio HUH7.0-soluissa lisääntyi myös nopeasti alkuvaiheen pimennysvaiheen jälkeen, mutta saavutti tasotason 24 tuntia injektion jälkeen solunsisäisessä osastossa 2 × 103 TCID50/ml:ssä ja väheni 66 tuntia injektion jälkeen. Virusta ei havaittu infektoitujen HUH7-solujen supernatantissa ennen kuin 36 tuntia inokulaation jälkeen, ja sen titterit olivat alhaisemmat kaikissa aikapisteissä (kuva 4). HUH7.0-soluissa ei koskaan havaittu suurta CPE:tä. Nämä tulokset ovat johdonmukaisia aiempien SARS-CoV:tä ja MERS-CoV:tä koskevien raporttien kanssa, jotka viittasivat zoonoottisten CoV-kantojen samanlaiseen replikaatiodynamiikkaan (23,24).
