Results
Figure 1
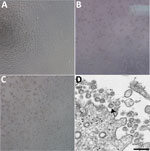
Figure 1. Efekt cytopatyczny wywołany przez koronawirusa 2 zespołu ostrej ciężkiej niewydolności oddechowej od pacjenta z chorobą koronawirusową, Stany Zjednoczone, 2020 r. A-C) Mikroskopia kontrastu fazowego monowarstw komórek Vero w 3 dniu po inokulacji: A) Mock,…
W dniu 22 stycznia 2020 r. w stanie Waszyngton zidentyfikowano pacjenta z potwierdzoną chorobą COVID-19. CPE nie zaobserwowano w komórkach zakażonych metodą mock (Figura 1, panel A). Wartości progu cyklu (Ct) wynosiły 18-20 dla próbek NP i 21-22 dla próbek OP (1). Pozytywne próbki kliniczne zostały podzielone na mniejsze części i ponownie zamrożone i zaszczepione do hodowli komórkowej 22 stycznia 2020 roku. Zaobserwowaliśmy CPE 2 dni po inokulacji i zebraliśmy lizat wirusowy w 3 dniu po inokulacji (Figura 1, panele B, C). Użyliśmy 50 μL lizatów wirusowych z pasażu 1 do ekstrakcji kwasu nukleinowego w celu potwierdzenia obecności SARS-CoV-2 za pomocą testu diagnostyki molekularnej CDC (1). Wartości Ct z 3 ekstrakcji kwasu nukleinowego wynosiły 16,0-17,1 dla części 1 nukleokapsydu, 15,9-17,1 dla części 2 nukleokapsydu i 16,2-17,3 dla części 3 nukleokapsydu, co potwierdziło izolację SARS-CoV-2 (Ct <40 jest uważane za wynik pozytywny). Testowaliśmy również ekstrakty na obecność 33 dodatkowych patogenów układu oddechowego przy użyciu testu Fast Track 33 Assay. Żadne inne patogeny nie zostały wykryte. Tożsamość została dodatkowo potwierdzona przez mikroskopię elektronową cienkiego przekroju (Figura 1, panel D). Zaobserwowaliśmy morfologię i morfogenezę charakterystyczną dla koronawirusów.
Użyliśmy izolatów z pierwszego przejścia OP i próbki NP do sekwencjonowania całego genomu. Genomy z okazu NP (GenBank accession MT020880) i okazu OP (GenBank accession no. MT020881) wykazały 100% identyczność ze sobą. Izolaty wykazały również 100% identyczność z odpowiadającym im preparatem klinicznym (GenBank accession no. MN985325).
Po drugim pasażu nie hodowaliśmy osobno próbek OP i NP. Izolat wirusa pasażowaliśmy jeszcze 2 razy w komórkach Vero CCL-81 i miareczkowaliśmy określając 50% dawkę zakaźną w hodowli tkankowej (TCID50). Miana wynosiły 8,65 × 106 TCID50/mL dla trzeciego pasażu i 7,65 × 106 TCID50/mL dla czwartego pasażu.
Pasażowaliśmy tego wirusa w nieobecności trypsyny. Sekwencja białka kolca wirusa SARS-CoV-2 zawiera wstawkę RRAR w interfejsie S1-S2, która może być rozszczepiona przez furynę (16). Wysoce patogenne wirusy ptasiej grypy mają wysoce podstawowe miejsca rozszczepienia furyn w interfejsie białka hemaglutyniny HA1-HA2, które umożliwiają wewnątrzkomórkowe dojrzewanie wirionów i bardziej wydajną replikację wirusową (17). Insercja RRAR w SARS-CoV-2 może pełnić podobną funkcję.
Rysunek 2
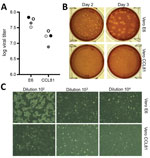
Rysunek 2. Rozmnażanie wirusa i oznaczanie ilościowe koronawirusa 2 zespołu ostrej ciężkiej niewydolności oddechowej od pacjenta z chorobą koronawirusową, Stany Zjednoczone, 2020 r. A) Dwa zapasy 4 pasażu wirusa (czarne i szare okręgi) zostały określone ilościowo…
Następnie wygenerowaliśmy zapas czwartego pasażu SARS-CoV-2 na komórkach VeroE6, innej linii komórek nerek płodowej małpy rhesus. Sekwencjonowaliśmy wirusowe RNA z czwartego pasażu SARS-CoV-2 i potwierdziliśmy, że nie posiada ono żadnych mutacji nukleotydowych w porównaniu z oryginalną sekwencją referencyjną (GenBank nr akcesyjny MN985325). Stwierdzono, że SARS-CoV dobrze rośnie na komórkach VeroE6, a MERS-CoV na komórkach Vero CCL81 (18,19). W celu ustanowienia testu płytkowego i określenia preferowanego typu komórek Vero do oznaczania ilościowego, miareczkowaliśmy nasz materiał wyjściowy z pasażu 4 na komórkach VeroE6 i VeroCCL81. Po zakażeniu serią rozcieńczeń, SARS-CoV-2 replikował się w obu typach komórek Vero; jednak miana wirusów były nieco wyższe w komórkach VeroE6 niż w komórkach Vero CCL81 (Figura 2, panel A). Ponadto blaszki były bardziej wyraźne i widoczne na komórkach Vero E6 (Figura 2, panel B). Już po 2 dniach postinokulacji komórki VeroE6 wytworzyły wyraźne płytki widoczne przez barwienie czerwienią obojętną. W przeciwieństwie do tego, komórki Vero CCL81 wytworzyły mniej wyraźne płytki, które najłatwiej było oznaczyć ilościowo przez barwienie czerwienią obojętną 3 dni po inokulacji. Na pojedynczych monowarstwach płytek, infekcja SARS-CoV-2 komórek Vero E6 wytworzyła CPE z obszarami usuwania komórek (Figura 2, panel C). W przeciwieństwie do tego, komórki Vero CCL81 miały obszary martwych komórek, które połączyły się, tworząc płytki, ale komórki nie uległy oczyszczeniu. Łącznie, wyniki te sugerują, że komórki VeroE6 mogą być najlepszym wyborem do amplifikacji i kwantyfikacji, ale oba typy komórek Vero wspierają amplifikację i replikację SARS-CoV-2.
Rysunek 3
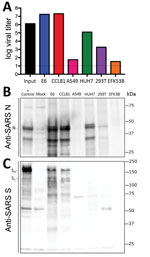
Rysunek 3. Linie komórkowe od pacjenta z chorobą koronawirusową, Stany Zjednoczone, 2020, wrażliwe na koronawirus SARS 2 (SARS-CoV-2). Linie komórkowe zostały zakażone z wysoką wielokrotnością infekcji (> 5), przemyte po adsorpcji, i…
Ponieważ badania zostały zainicjowane w celu zbadania i odpowiedzi na SARS-CoV-2, potrzebne są informacje o liniach komórkowych i typach podatnych na infekcję. Dlatego zbadaliśmy zdolność SARS-CoV-2 do infekowania i replikacji w kilku powszechnych liniach komórkowych naczelnych i ludzkich, w tym ludzkich komórkach gruczolakoraka (A549), ludzkich komórkach wątroby (HUH7.0) i ludzkich komórkach nerki embrionalnej (HEK-293T), oprócz komórek Vero E6 i Vero CCL81. Zbadaliśmy również dostępną linię komórkową nerki nietoperza brunatnego (EFK3B) pod kątem zdolności do replikacji SARS-CoV-2. Każda linia komórkowa była inokulowana przy wysokiej wielokrotności infekcji i badana 24 h po infekcji (Figura 3, panel A). Nie zaobserwowano CPE w żadnej z linii komórkowych, z wyjątkiem komórek Vero, które wzrosły do >107 PFU w 24 h po zakażeniu. W przeciwieństwie do tego, komórki HUH7.0 i 293T wykazywały tylko umiarkowaną replikację wirusową, a komórki A549 były niekompatybilne z infekcją SARS-CoV-2. Wyniki te są zgodne z wcześniejszymi ustaleniami dotyczącymi wrażliwości na SARS-CoV i sugerują, że inne powszechnie stosowane systemy hodowlane, w tym MDCK, HeLa, HEP-2, komórki MRC-5 i jaja zarodkowe, prawdopodobnie nie wspierają replikacji SARS-CoV-2 (20-22). Ponadto, SARS-CoV-2 nie replikował się w komórkach nietoperzy EFK3B, które są wrażliwe na MERS-CoV. Łącznie wyniki te wskazują, że SARS-CoV-2 utrzymuje podobny profil do SARS-CoV pod względem podatnych linii komórkowych.
Po ustanowieniu silnej infekcji SARS-CoV-2 w kilku typach komórek, następnie oceniliśmy reaktywność krzyżową przeciwciał przeciwko SARS-CoV-2. Lizaty komórkowe z zainfekowanych linii komórkowych zostały poddane analizie białkowej; stwierdziliśmy, że surowica poliklonalna przeciwko białku szpiku SARS-CoV i białkom nukleokapsydu rozpoznaje SARS-CoV-2 (Figura 3, panele B, C). Białko nukleokapsydu, które jest wysoce konserwowane w całej rodzinie grupy 2B, zachowuje >90% identyczności aminokwasów między SARS-CoV i SARS-CoV-2. Zgodnie z wynikami replikacji (Figura 3, panel A), SARS-CoV-2 wykazał silne białko nukleokapsydu w obu typach komórek Vero, mniej białka w komórkach HUH7.0 i 293T oraz minimalne białko w komórkach A549 i EFK3B (Figura 3, panel B). Przeciwciało przeciw białku spike SARS-CoV rozpoznawało również białko spike SARS-CoV-2, wskazując na reaktywność krzyżową (Figura 3, panel C). Zgodnie z SARS CoV, zaobserwowano kilka rozszczepionych i nie rozszczepionych form białka kolców SARS-CoV-2. Wzór rozszczepienia pozytywnej kontroli kolców SARS z komórek Calu3, linii komórek oddechowych, różni się nieznacznie i może wskazywać na różnice między proteolitycznym rozszczepieniem białek kolców między tymi dwoma wirusami z powodu przewidywanego wprowadzenia miejsca rozszczepienia furyn w SARS-CoV-2 (16). Jednakże różnice w typie komórek i warunkach komplikują tę interpretację i wskazują na potrzebę dalszych badań w równoważnych systemach. Ogólnie rzecz biorąc, dane dotyczące ekspresji białek z przeciwciał przeciwko nukleokapsydowi SARS-CoV i białku spike rekapitulują wyniki replikacji i wskazują, że odczynniki SARS-CoV mogą być używane do charakteryzowania infekcji SARS-CoV-2.
Rysunek 4
Rysunek 4. Wieloetapowa krzywa wzrostu dla koronawirusa 2 zespołu ostrej ciężkiej niewydolności oddechowej od pacjenta z chorobą koronawirusową, Stany Zjednoczone, 2020 r. Komórki Vero CCL81 (czarny) i HUH7.0 (zielony) zostały zakażone przy krotności…
W końcu oceniliśmy kinetykę replikacji SARS-CoV-2 w wielostopniowej krzywej wzrostu. W skrócie, zainfekowaliśmy komórki Vero CCL-81 i HUH7.0 komórkami SARS-CoV-2 przy niskiej krotności zakażenia (0,1) i ocenialiśmy replikację wirusową co 6 h przez 72 h po inokulacji, z oddzielnymi zbiorami w przedziałach związanych z komórkami i supernatantu (Figura 4). Podobnie do SARS-CoV, SARS-CoV-2 replikował się szybko w komórkach Vero po początkowej fazie zaćmienia, osiągając 105 TCID50/mL w 24 h po zakażeniu i osiągając szczyt przy >106 TCID50/mL. Zaobserwowaliśmy podobne miana w kompartmencie związanym z komórkami i supernatancie, co wskazuje na wydajne wydalanie. Pomimo szczytowego miana wirusa w 48 h po inokulacji, główny CPE nie był obserwowany do 60 h po inokulacji i osiągnął szczyt w 72 h po inokulacji, wskazując, że zainfekowane monowarstwy powinny być zbierane przed zaobserwowaniem szczytowego CPE. Replikacja w komórkach HUH7.0 również szybko wzrastała po początkowej fazie zaćmienia, ale osiągnęła plateau w 24 h po inokulacji w przedziale wewnątrzkomórkowym przy 2 × 103 TCID50/mL i spadła po 66 h po inokulacji. Wirus nie był wykrywany w supernatancie zakażonych komórek HUH7 do 36 h po inokulacji i wykazywał niższe miana we wszystkich punktach czasowych (Rysunek 4). Większy CPE nigdy nie był obserwowany w komórkach HUH7.0. Wyniki te są zgodne z wcześniejszymi doniesieniami dotyczącymi SARS-CoV i MERS-CoV, które sugerowały podobną dynamikę replikacji pomiędzy odzwierzęcymi szczepami CoV (23,24).
