Resultados
Figura 1
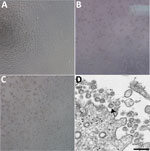
Figura 1. Efecto citopático causado por el coronavirus del síndrome respiratorio agudo grave 2 de un paciente con enfermedad por coronavirus, Estados Unidos, 2020. A-C) Microscopía de contraste de fase de monocapas de células Vero a los 3 días postinoculación: A) Simulacro,…
Se identificó un paciente con COVID-19 confirmado en el estado de Washington el 22 de enero de 2020. No se observó CPE en las células infectadas de prueba (Figura 1, panel A). Los valores del umbral de ciclo (Ct) fueron de 18-20 para los especímenes NP y de 21-22 para los especímenes OP (1). Las muestras clínicas positivas se alicuotaron y se volvieron a congelar en el cultivo celular el 22 de enero de 2020. Observamos el CPE 2 días después de la inoculación y cosechamos el lisado viral el día 3 después de la inoculación (Figura 1, paneles B, C). Utilizamos 50 μL de lisados virales de paso 1 para la extracción de ácido nucleico para confirmar la presencia de SARS-CoV-2 mediante el ensayo de diagnóstico molecular de los CDC (1). Los valores de Ct de 3 extracciones de ácido nucleico fueron de 16,0-17,1 para la porción 1 de la nucleocápside, de 15,9-17,1 para la porción 2 de la nucleocápside y de 16,2-17,3 para la porción 3 de la nucleocápside, lo que confirmó el aislamiento del SARS-CoV-2 (Ct <40 se considera un resultado positivo). También analizamos los extractos en busca de otros 33 patógenos respiratorios diferentes utilizando el ensayo Fast Track 33. No se detectó ningún otro patógeno. La identidad fue apoyada adicionalmente por la microscopía electrónica de sección fina (Figura 1, panel D). Observamos una morfología y morfogénesis características de los coronavirus.
Utilizamos aislados del primer pasaje de un espécimen OP y un espécimen NP para la secuenciación del genoma completo. Los genomas del espécimen NP (acceso al GenBank MT020880) y del espécimen OP (acceso al GenBank nº MT020881) mostraron un 100% de identidad entre sí. Los aislados también mostraron un 100% de identidad con el espécimen clínico correspondiente (número de acceso del GenBank MN985325).
Después del segundo pasaje, no cultivamos los especímenes OP y NP por separado. Pasamos el virus aislado 2 veces más en células Vero CCL-81 y titulamos determinando la dosis infecciosa de cultivo de tejidos del 50% (TCID50). Los títulos fueron de 8,65 × 106 DICT50/mL para el tercer pasaje y de 7,65 × 106 DICT50/mL para el cuarto pasaje.
Pasamos este virus en ausencia de tripsina. La secuencia de la proteína de espiga del SARS-CoV-2 tiene una inserción RRAR en la interfaz S1-S2 que podría ser escindida por la furina (16). Los virus de la gripe aviar altamente patógenos tienen sitios de escisión de furina altamente básicos en la interfaz de la proteína hemaglutinina HA1-HA2 que permiten la maduración intracelular de los viriones y una replicación viral más eficiente (17). La inserción de RRAR en el SARS-CoV-2 podría cumplir una función similar.
Figura 2
Figura 2. Propagación viral y cuantificación del coronavirus del síndrome respiratorio agudo severo 2 de un paciente con enfermedad por coronavirus, Estados Unidos, 2020. A) Se cuantificaron dos stocks de virus de paso 4 (círculos negros y grises)…
Posteriormente generamos un stock de cuarto paso de SARS-CoV-2 en células VeroE6, otra línea celular de riñón de mono rhesus fetal. Secuenciamos el ARN viral del stock de SARS-CoV-2 del cuarto pasaje y confirmamos que no tenía mutaciones nucleotídicas en comparación con la secuencia de referencia original (número de acceso al GenBank MN985325). Se ha comprobado que el SARS-CoV crece bien en células VeroE6 y el MERS-CoV en células Vero CCL81 (18,19). Para establecer un ensayo de placa y determinar el tipo de célula Vero preferido para la cuantificación, titulamos nuestro stock de paso 4 en células VeroE6 y VeroCCL81. Tras la infección con una serie de diluciones, el SARS-CoV-2 se replicó en ambos tipos de células Vero; sin embargo, los títulos virales fueron ligeramente superiores en las células VeroE6 que en las células Vero CCL81 (Figura 2, panel A). Además, las placas eran más definidas y visibles en las células Vero E6 (Figura 2, panel B). A partir de los 2 días posteriores a la inoculación, las células VeroE6 produjeron placas distintas visibles mediante tinción con rojo neutro. Por el contrario, las células Vero CCL81 produjeron placas menos claras y fueron más fácilmente cuantificables mediante tinción con rojo neutro a los 3 días postinoculación. En las monocapas de placas individuales, la infección por el SARS-CoV-2 de las células Vero E6 produjo CPE con áreas de eliminación de células (Figura 2, panel C). Por el contrario, las células Vero CCL81 tenían áreas de células muertas que se habían fusionado para formar placas, pero las células no se despejaron. En conjunto, estos resultados sugieren que las células VeroE6 podrían ser la mejor opción para la amplificación y cuantificación, pero ambos tipos de células Vero admiten la amplificación y replicación del SARS-CoV-2.
Figura 3
Figura 3. Líneas celulares de paciente con enfermedad por coronavirus, Estados Unidos, 2020, susceptibles al coronavirus 2 del SARS (SARS-CoV-2). Las líneas celulares se infectaron con una alta multiplicidad de infección (>5), se lavaron después de la adsorción y…
Debido a que se ha iniciado la investigación para estudiar y responder al SARS-CoV-2, se necesita información sobre las líneas y tipos celulares susceptibles a la infección. Por lo tanto, examinamos la capacidad del SARS-CoV-2 para infectar y replicarse en varias líneas celulares comunes de primates y humanos, incluyendo células de adenocarcinoma humano (A549), células de hígado humano (HUH7.0) y células de riñón embrionario humano (HEK-293T), además de las células Vero E6 y Vero CCL81. También examinamos una línea celular de riñón de murciélago grande disponible (EFK3B) para comprobar la capacidad de replicación del SARS-CoV-2. Cada línea celular se inoculó con una alta multiplicidad de infección y se examinó 24 h después de la infección (Figura 3, panel A). No se observó CPE en ninguna de las líneas celulares, excepto en las células Vero, que crecieron hasta >107 PFU a las 24 h de la infección. Por el contrario, las células HUH7.0 y 293T sólo mostraron una modesta replicación viral, y las células A549 fueron incompatibles con la infección por SARS-CoV-2. Estos resultados son coherentes con los hallazgos anteriores sobre la susceptibilidad al SARS-CoV y sugieren que es poco probable que otros sistemas de cultivo comunes, como las células MDCK, HeLa, HEP-2, MRC-5 y los huevos embrionados, soporten la replicación del SARS-CoV-2 (20-22). Además, el SARS-CoV-2 no se replicó en células EFK3B de murciélago, que son susceptibles al MERS-CoV. En conjunto, los resultados indican que el SARS-CoV-2 mantiene un perfil similar al SARS-CoV en términos de líneas celulares susceptibles.
Habiendo establecido una infección robusta con el SARS-CoV-2 en varios tipos celulares, evaluamos a continuación la reactividad cruzada de los anticuerpos contra el SARS-CoV-2. Se analizaron los lisados celulares de las líneas celulares infectadas para el análisis de las proteínas; descubrimos que el suero policlonal contra la proteína de la espiga del SARS-CoV y las proteínas de la nucleocápside reconocen el SARS-CoV-2 (Figura 3, paneles B, C). La proteína de la nucleocápside, que está muy conservada en toda la familia del grupo 2B, mantiene una identidad de aminoácidos de >90% entre el SARS-CoV y el SARS-CoV-2. En consonancia con los resultados de la replicación (Figura 3, panel A), el SARS-CoV-2 mostró una sólida proteína de nucleocápside en ambos tipos de células Vero, menos proteína en las células HUH7.0 y 293T, y una proteína mínima en las células A549 y EFK3B (Figura 3, panel B). El anticuerpo de la proteína de espiga del SARS-CoV también reconoció la proteína de espiga del SARS-CoV-2, lo que indica una reactividad cruzada (Figura 3, panel C). En consonancia con el CoV del SRAS, se observaron varias formas escindidas y no escindidas de la proteína de espiga del SRAS-CoV-2. El patrón de clivaje del control positivo de la espiga del SARS de las células Calu3, una línea celular respiratoria, varía ligeramente y podría indicar diferencias entre el clivaje proteolítico de las proteínas de la espiga entre los 2 virus debido a la inserción prevista de un sitio de clivaje de furina en el SARS-CoV-2 (16). Sin embargo, las diferencias en el tipo de célula y las condiciones complican esta interpretación e indican la necesidad de realizar más estudios en sistemas equivalentes. En general, los datos de expresión proteica de los anticuerpos contra la nucleocápside y la proteína de espiga del SARS-CoV recapitulan los resultados de la replicación e indican que los reactivos del SARS-CoV pueden utilizarse para caracterizar la infección por el SARS-CoV-2.
Figura 4
Figura 4. Curva de crecimiento multipaso del coronavirus del síndrome respiratorio agudo severo 2 de un paciente con enfermedad por coronavirus, Estados Unidos, 2020. Las células Vero CCL81 (negro) y HUH7.0 (verde) se infectaron a una multiplicidad de…
Por último, evaluamos la cinética de replicación del SARS-CoV-2 en una curva de crecimiento de varios pasos. En resumen, infectamos células Vero CCL-81 y HUH7.0 con SARS-CoV-2 a una multiplicidad de infección baja (0,1) y evaluamos la replicación viral cada 6 h durante 72 h después de la inoculación, con cosechas separadas en los compartimentos asociado a la célula y sobrenadante (Figura 4). Al igual que el SARS-CoV, el SARS-CoV-2 se replicó rápidamente en las células Vero después de una fase inicial de eclipse, alcanzando 105 TCID50/mL a las 24 h de la postinfección y alcanzando un máximo de >106 TCID50/mL. Observamos títulos similares en los compartimentos asociados a las células y en el sobrenadante, lo que indica una salida eficiente. A pesar de los títulos virales máximos a las 48 horas después de la inoculación, no se observó un CPE importante hasta las 60 horas después de la inoculación y alcanzó el máximo a las 72 horas después de la inoculación, lo que indica que las monocapas infectadas deben cosecharse antes de que se observe el CPE máximo. La replicación en las células HUH7.0 también aumentó rápidamente tras una fase inicial de eclipse, pero se estabilizó a las 24 h de la postinoculación en el compartimento intracelular a 2 × 103 DICT50/mL y disminuyó tras 66 h de la postinoculación. El virus no se detectó en el sobrenadante de las células HUH7 infectadas hasta las 36 h postinoculación y mostró títulos más bajos en todos los puntos temporales (Figura 4). En las células HUH7.0 nunca se observó un CPE importante. Estos resultados son coherentes con informes anteriores sobre el SARS-CoV y el MERS-CoV, que sugieren una dinámica de replicación similar entre las cepas zoonóticas del CoV (23,24).
