Résultats
Figure 1
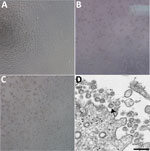
Figure 1. Effet cytopathique provoqué par le coronavirus 2 du syndrome respiratoire aigu sévère chez un patient atteint de coronavirus, États-Unis, 2020. A-C) Microscopie à contraste de phase de monocouches de cellules Vero à 3 jours après l’inoculation : A) Mock,…
Un patient a été identifié avec COVID-19 confirmé dans l’État de Washington le 22 janvier 2020. L’ECP n’a pas été observé dans les cellules infectées par un simulacre (figure 1, panneau A). Les valeurs de seuil de cycle (Ct) étaient de 18-20 pour les spécimens NP et de 21-22 pour les spécimens OP (1). Les spécimens cliniques positifs ont été aliquotés et recongelés pour être inoculés dans une culture cellulaire le 22 janvier 2020. Nous avons observé l’ECP 2 jours après l’inoculation et récolté le lysat viral le troisième jour après l’inoculation (figure 1, panneaux B, C). Nous avons utilisé 50 μL de lysats viraux de passage 1 pour l’extraction de l’acide nucléique afin de confirmer la présence du SRAS-CoV-2 en utilisant le test de diagnostic moléculaire du CDC (1). Les valeurs Ct de 3 extractions d’acide nucléique étaient de 16,0-17,1 pour la partie 1 de la nucléocapside, de 15,9-17,1 pour la partie 2 de la nucléocapside et de 16,2-17,3 pour la partie 3 de la nucléocapside, ce qui a confirmé l’isolement du SARS-CoV-2 (Ct <40 est considéré comme un résultat positif). Nous avons également testé les extraits pour 33 autres agents pathogènes respiratoires différents en utilisant le test Fast Track 33. Aucun autre agent pathogène n’a été détecté. L’identité a également été confirmée par la microscopie électronique en coupe fine (figure 1, panneau D). Nous avons observé une morphologie et une morphogenèse caractéristiques des coronavirus.
Nous avons utilisé des isolats du premier passage d’un spécimen OP et d’un spécimen NP pour le séquençage du génome entier. Les génomes du spécimen NP (accession GenBank MT020880) et du spécimen OP (accession GenBank n° MT020881) ont montré une identité de 100% entre eux. Les isolats ont également montré une identité de 100% avec le spécimen clinique correspondant (GenBank accession no. MN985325).
Après le deuxième passage, nous n’avons pas cultivé séparément les spécimens OP et NP. Nous avons fait passer l’isolat viral 2 fois de plus dans des cellules Vero CCL-81 et nous avons titré en déterminant la dose infectieuse de 50 % de la culture tissulaire (TCID50). Les titres étaient de 8,65 × 106 TCID50/mL pour le troisième passage et de 7,65 × 106 TCID50/mL pour le quatrième passage.
Nous avons fait passer ce virus en l’absence de trypsine. La séquence de la protéine de pointe du SARS-CoV-2 comporte une insertion RRAR à l’interface S1-S2 qui pourrait être clivée par la furine (16). Les virus de la grippe aviaire hautement pathogènes possèdent des sites de clivage par la furine à l’interface de la protéine hémagglutinine HA1-HA2 qui permettent la maturation intracellulaire des virions et une réplication virale plus efficace (17). L’insertion RRAR dans le SRAS-CoV-2 pourrait avoir une fonction similaire.
Figure 2
Figure 2. Propagation virale et quantification du coronavirus 2 du syndrome respiratoire aigu sévère à partir d’un patient atteint de coronavirus, États-Unis, 2020. A) Deux stocks de virus de quatrième passage (cercles noirs et gris) ont été quantifiés…
Nous avons ensuite généré un stock de quatrième passage de SARS-CoV-2 sur des cellules VeroE6, une autre lignée cellulaire de rein de singe rhésus fœtal. Nous avons séquencé l’ARN viral du stock de passage 4 du SRAS-CoV-2 et confirmé qu’il ne présentait aucune mutation nucléotidique par rapport à la séquence de référence originale (numéro d’accession GenBank MN985325). On a constaté que le SARS-CoV se développe bien sur les cellules VeroE6 et le MERS-CoV sur les cellules Vero CCL81 (18,19). Afin d’établir un test de plaque et de déterminer le type de cellule Vero à privilégier pour la quantification, nous avons titré notre stock de passage 4 sur des cellules VeroE6 et VeroCCL81. Après infection par une série de dilutions, le SRAS-CoV-2 s’est répliqué dans les deux types de cellules Vero ; toutefois, les titres viraux étaient légèrement plus élevés dans les cellules VeroE6 que dans les cellules Vero CCL81 (figure 2, panneau A). En outre, les plaques étaient plus distinctes et visibles sur les cellules Vero E6 (figure 2, panneau B). Dès le deuxième jour après l’inoculation, les cellules VeroE6 ont produit des plaques distinctes visibles par coloration au rouge neutre. En revanche, les cellules Vero CCL81 ont produit des plaques moins nettes, qui étaient plus facilement quantifiables par coloration au rouge neutre 3 jours après l’inoculation. Sur les monocouches de plaques individuelles, l’infection par le SRAS-CoV-2 des cellules Vero E6 a produit un ECP avec des zones de clairance cellulaire (figure 2, panneau C). En revanche, les cellules Vero CCL81 présentaient des zones de cellules mortes qui avaient fusionné pour former des plaques, mais les cellules n’ont pas disparu. Ensemble, ces résultats suggèrent que les cellules VeroE6 pourraient être le meilleur choix pour l’amplification et la quantification, mais les deux types de cellules Vero supportent l’amplification et la réplication du SRAS-CoV-2.
Figure 3
Figure 3. Lignées cellulaires provenant d’un patient atteint de coronavirus, États-Unis, 2020, sensibles au coronavirus 2 du SRAS (SARS-CoV-2). Les lignées cellulaires ont été infectées avec une multiplicité d’infection élevée (>5), lavées après adsorption, et…
Parce que des recherches ont été initiées pour étudier et répondre au SARS-CoV-2, des informations sur les lignées cellulaires et les types sensibles à l’infection sont nécessaires. Par conséquent, nous avons examiné la capacité du SRAS-CoV-2 à infecter et à se répliquer dans plusieurs lignées cellulaires humaines et de primates communes, y compris les cellules d’adénocarcinome humain (A549), les cellules de foie humain (HUH7.0) et les cellules de rein embryonnaire humain (HEK-293T), en plus des cellules Vero E6 et Vero CCL81. Nous avons également examiné la capacité de réplication du virus SRAS-CoV-2 d’une lignée cellulaire de rein de grande chauve-souris brune disponible (EFK3B). Chaque lignée cellulaire a été inoculée à une multiplicité d’infection élevée et examinée 24 heures après l’infection (figure 3, panneau A). Aucun ECP n’a été observé dans aucune des lignées cellulaires, sauf dans les cellules Vero, dont la croissance était de >107 UFP à 24 h post-infection. En revanche, les cellules HUH7.0 et 293T n’ont présenté qu’une réplication virale modeste, et les cellules A549 étaient incompatibles avec l’infection par le SRAS-CoV-2. Ces résultats sont conformes aux constatations antérieures sur la sensibilité au CoV SRAS et donnent à penser que d’autres systèmes de culture courants, notamment les cellules MDCK, HeLa, HEP-2, MRC-5 et les œufs embryonnés, sont peu susceptibles de favoriser la réplication du CoV SRAS-2 (20-22). De plus, le SRAS-CoV-2 ne s’est pas répliqué dans les cellules EFK3B de chauve-souris, qui sont sensibles au MERS-CoV. Ces résultats indiquent que le SARS-CoV-2 présente un profil similaire à celui du SARS-CoV en ce qui concerne les lignées cellulaires sensibles.
Après avoir établi une infection robuste par le SARS-CoV-2 dans plusieurs types de cellules, nous avons ensuite évalué la réactivité croisée des anticorps dirigés contre le SARS-CoV-2. Des lysats cellulaires provenant de lignées cellulaires infectées ont été soumis à une analyse protéique ; nous avons constaté que les sérums polyclonaux dirigés contre la protéine de pointe du SRAS-CoV et les protéines de la nucléocapside reconnaissent le SRAS-CoV-2 (figure 3, panneaux B, C). La protéine de la nucléocapside, qui est hautement conservée dans toute la famille du groupe 2B, conserve une identité d’acides aminés de >90 % entre le SARS-CoV et le SARS-CoV-2. Conformément aux résultats de la réplication (figure 3, panneau A), le SARS-CoV-2 a présenté une protéine de nucléocapside robuste dans les deux types de cellules Vero, une protéine moindre dans les cellules HUH7.0 et 293T, et une protéine minimale dans les cellules A549 et EFK3B (figure 3, panneau B). L’anticorps de la protéine de pointe du SRAS-CoV a également reconnu la protéine de pointe du SRAS-CoV-2, ce qui indique une réactivité croisée (figure 3, panneau C). Conformément au CoV du SRAS, plusieurs formes clivées et non clivées de la protéine de pointe du CoV-2 du SRAS ont été observées. Le schéma de clivage du contrôle positif de la protéine spike du SRAS provenant des cellules Calu3, une lignée cellulaire respiratoire, varie légèrement et pourrait indiquer des différences entre le clivage protéolytique des protéines spike entre les 2 virus en raison de l’insertion prédite d’un site de clivage de la furine dans le SARS-CoV-2 (16). Cependant, les différences dans le type de cellule et les conditions compliquent cette interprétation et indiquent la nécessité d’une étude plus approfondie dans des systèmes équivalents. Dans l’ensemble, les données d’expression protéique des anticorps de la nucléocapside et de la protéine de pointe du SARS-CoV récapitulent les résultats de la réplication et indiquent que les réactifs du SARS-CoV peuvent être utilisés pour caractériser l’infection par le SARS-CoV-2.
Figure 4
Figure 4. Courbe de croissance multi-étapes du coronavirus 2 du syndrome respiratoire aigu sévère provenant d’un patient atteint de coronavirus, États-Unis, 2020. Les cellules Vero CCL81 (noir) et HUH7.0 (vert) ont été infectées à une multiplicité de…
Enfin, nous avons évalué la cinétique de réplication du SARS-CoV-2 dans une courbe de croissance à plusieurs étapes. En bref, nous avons infecté des cellules Vero CCL-81 et HUH7.0 avec le SARS-CoV-2 à une faible multiplicité d’infection (0,1) et évalué la réplication virale toutes les 6 heures pendant 72 heures après l’inoculation, avec des récoltes séparées dans les compartiments associés aux cellules et dans le surnageant (figure 4). Comme pour le SARS-CoV, le SARS-CoV-2 s’est répliqué rapidement dans les cellules Vero après une phase initiale d’éclipse, atteignant 105 DICT50/mL 24 heures après l’infection et culminant à >106 DICT50/mL. Nous avons observé des titres similaires dans les compartiments associés aux cellules et dans le surnageant, ce qui indique une sortie efficace. Malgré des titres viraux maximums 48 heures après l’inoculation, un ECP majeur n’a été observé que 60 heures après l’inoculation et a atteint son maximum 72 heures après l’inoculation, ce qui indique que les monocouches infectées doivent être récoltées avant que l’ECP ne soit observé. La réplication dans les cellules HUH7.0 a également augmenté rapidement après une phase initiale d’éclipse, mais a atteint un plateau 24 h après l’inoculation dans le compartiment intracellulaire à 2 × 103 TCID50/mL et a diminué après 66 h après l’inoculation. Le virus n’a pas été détecté dans le surnageant des cellules HUH7 infectées jusqu’à 36 heures après l’inoculation et a présenté des titres plus faibles à tous les moments (figure 4). Un ECP majeur n’a jamais été observé dans les cellules HUH7.0. Ces résultats sont conformes aux rapports précédents concernant le SARS-CoV et le MERS-CoV, qui suggéraient une dynamique de réplication similaire entre les souches de CoV zoonotiques (23,24).
