Ergebnisse
Abbildung 1
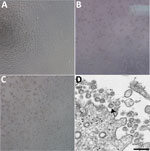
Abbildung 1. Zytopathischer Effekt, verursacht durch das schwere akute respiratorische Syndrom Coronavirus 2 bei einem Patienten mit Coronavirus-Erkrankung, Vereinigte Staaten, 2020. A-C) Phasenkontrastmikroskopie von Vero-Zellmonolayern 3 Tage nach der Inokulation: A) Mock,…
Ein Patient wurde am 22. Januar 2020 mit bestätigtem COVID-19 im Bundesstaat Washington identifiziert. In mock-infizierten Zellen wurde kein CPE beobachtet (Abbildung 1, Tafel A). Die Zyklusschwellenwerte (Ct) lagen bei 18-20 für NP-Proben und 21-22 für OP-Proben (1). Die positiven klinischen Proben wurden aliquotiert und am 22. Januar 2020 wieder eingefroren und in Zellkulturen beimpft. Wir beobachteten den CPE 2 Tage nach der Inokulation und ernteten das virale Lysat an Tag 3 nach der Inokulation (Abbildung 1, Felder B, C). Wir verwendeten 50 μl der viralen Lysate der Passage 1 für die Nukleinsäureextraktion, um das Vorhandensein von SARS-CoV-2 mit Hilfe des molekulardiagnostischen CDC-Assays zu bestätigen (1). Die Ct-Werte von drei Nukleinsäureextraktionen betrugen 16,0-17,1 für den Nukleokapsidteil 1, 15,9-17,1 für den Nukleokapsidteil 2 und 16,2-17,3 für den Nukleokapsidteil 3, was die Isolierung von SARS-CoV-2 bestätigte (Ct <40 gilt als positives Ergebnis). Mit dem Fast Track 33 Assay wurden die Extrakte auch auf 33 weitere Atemwegserreger getestet. Es wurden keine weiteren Erreger nachgewiesen. Die Identität wurde zusätzlich durch Dünnschliff-Elektronenmikroskopie bestätigt (Abbildung 1, Tafel D). Wir beobachteten eine für Coronaviren charakteristische Morphologie und Morphogenese.
Wir verwendeten Isolate aus der ersten Passage einer OP- und einer NP-Probe für die Ganzgenomsequenzierung. Die Genome der NP-Probe (GenBank-Zugangsnummer MT020880) und der OP-Probe (GenBank-Zugangsnummer MT020881) waren zu 100 % identisch. Die Isolate wiesen auch eine 100%ige Identität mit der entsprechenden klinischen Probe (GenBank-Zugangsnummer MN985325) auf.
Nach der zweiten Passage kultivierten wir OP- und NP-Proben nicht mehr getrennt. Wir passagierten das Virusisolat zwei weitere Male in Vero CCL-81-Zellen und titrierten durch Bestimmung der 50%igen infektiösen Gewebekulturdosis (TCID50). Die Titer betrugen 8,65 × 106 TCID50/ml für die dritte Passage und 7,65 × 106 TCID50/ml für die vierte Passage.
Wir passagierten dieses Virus in Abwesenheit von Trypsin. Die Spike-Protein-Sequenz von SARS-CoV-2 hat eine RRAR-Insertion an der S1-S2-Schnittstelle, die von Furin gespalten werden könnte (16). Hochpathogene aviäre Influenzaviren haben hochgradig basische Furin-Spaltstellen an der Schnittstelle des Hämagglutinin-Proteins HA1-HA2, die eine intrazelluläre Reifung der Virionen und eine effizientere Virusreplikation ermöglichen (17). Die RRAR-Insertion in SARS-CoV-2 könnte eine ähnliche Funktion haben.
Abbildung 2
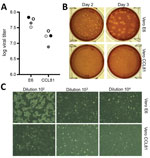
Abbildung 2. Virale Vermehrung und Quantifizierung des Coronavirus 2 des schweren akuten respiratorischen Syndroms aus einem Patienten mit Coronavirus-Erkrankung, Vereinigte Staaten, 2020. A) Zwei Viren der Passage 4 (schwarze und graue Kreise) wurden quantifiziert…
Anschließend erzeugten wir eine vierte Passage von SARS-CoV-2 auf VeroE6-Zellen, einer weiteren fötalen Rhesusaffen-Nierenzelllinie. Wir sequenzierten die virale RNA aus dem SARS-CoV-2-Passage-4-Stamm und bestätigten, dass sie im Vergleich zur ursprünglichen Referenzsequenz (GenBank-Zugangsnummer MN985325) keine Nukleotidmutationen aufweist. Es hat sich gezeigt, dass SARS-CoV gut auf VeroE6-Zellen und MERS-CoV auf Vero CCL81-Zellen wächst (18,19). Um einen Plaque-Assay zu etablieren und den bevorzugten Vero-Zelltyp für die Quantifizierung zu bestimmen, titerten wir unseren Passage-4-Stamm auf VeroE6- und VeroCCL81-Zellen. Nach der Infektion mit einer Verdünnungsreihe replizierte SARS-CoV-2 in beiden Vero-Zelltypen; die Virustiter waren jedoch in VeroE6-Zellen etwas höher als in Vero CCL81-Zellen (Abbildung 2, Tafel A). Darüber hinaus waren die Plaques auf Vero-E6-Zellen deutlicher ausgeprägt und sichtbar (Abbildung 2, Tafel B). Bereits 2 Tage nach der Inokulation bildeten VeroE6-Zellen deutliche Plaques, die durch Färbung mit Neutralrot sichtbar wurden. Im Gegensatz dazu bildeten Vero CCL81-Zellen weniger deutliche Plaques, die sich am einfachsten durch Färbung mit Neutralrot 3 Tage nach der Inokulation quantifizieren ließen. Auf den einzelnen Plaque-Monolayern führte die SARS-CoV-2-Infektion von Vero E6-Zellen zu CPE mit Bereichen, in denen sich die Zellen auflösten (Abbildung 2, Tafel C). Im Gegensatz dazu wiesen die Vero CCL81-Zellen Bereiche mit abgestorbenen Zellen auf, die zu Plaques verschmolzen waren, aber die Zellen lösten sich nicht auf. Zusammengenommen deuten diese Ergebnisse darauf hin, dass VeroE6-Zellen die beste Wahl für die Amplifikation und Quantifizierung sein könnten, aber beide Vero-Zelltypen unterstützen die Amplifikation und Replikation von SARS-CoV-2.
Abbildung 3
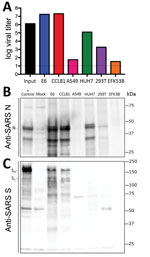
Abbildung 3. Zelllinien von Patienten mit Coronavirus-Erkrankung, Vereinigte Staaten, 2020, die für das SARS-Coronavirus 2 (SARS-CoV-2) empfänglich sind. Die Zelllinien wurden mit einer hohen Infektionsmultiplikation (>5) infiziert, nach der Adsorption gewaschen und…
Da die Forschung zur Untersuchung von und Reaktion auf SARS-CoV-2 eingeleitet wurde, werden Informationen über die für eine Infektion anfälligen Zelllinien und -typen benötigt. Daher haben wir die Fähigkeit von SARS-CoV-2 untersucht, mehrere gängige Primaten- und Humanzelllinien zu infizieren und zu vermehren, darunter menschliche Adenokarzinomzellen (A549), menschliche Leberzellen (HUH7.0) und menschliche embryonale Nierenzellen (HEK-293T) sowie Vero E6- und Vero CCL81-Zellen. Außerdem untersuchten wir eine verfügbare Nierenzelllinie der Großen Braunen Fledermaus (EFK3B) auf die Replikationsfähigkeit von SARS-CoV-2. Jede Zelllinie wurde mit einer hohen Infektionsmultiplikation inokuliert und 24 Stunden nach der Infektion untersucht (Abbildung 3, Tafel A). Bei keiner der Zelllinien wurde ein CPE beobachtet, außer bei Vero-Zellen, die 24 Stunden nach der Infektion auf >107 PFU anwuchsen. Im Gegensatz dazu zeigten HUH7.0- und 293T-Zellen nur eine bescheidene Virusreplikation, und A549-Zellen waren mit einer SARS-CoV-2-Infektion nicht kompatibel. Diese Ergebnisse stimmen mit früheren Erkenntnissen über die Anfälligkeit von SARS-CoV überein und deuten darauf hin, dass andere gängige Kultursysteme, darunter MDCK-, HeLa-, HEP-2-, MRC-5-Zellen und embryonierte Eier, die Replikation von SARS-CoV-2 wahrscheinlich nicht unterstützen (20-22). Darüber hinaus replizierte sich SARS-CoV-2 nicht in EFK3B-Zellen der Fledermaus, die für MERS-CoV empfänglich sind. Insgesamt deuten die Ergebnisse darauf hin, dass SARS-CoV-2 ein ähnliches Profil wie SARS-CoV in Bezug auf anfällige Zelllinien aufweist.
Nachdem wir eine robuste Infektion mit SARS-CoV-2 in verschiedenen Zelltypen festgestellt hatten, untersuchten wir als nächstes die Kreuzreaktivität von SARS-CoV-Antikörpern gegen SARS-CoV-2. Zelllysate von infizierten Zelllinien wurden für die Proteinanalyse untersucht. Es zeigte sich, dass polyklonale Serumproben gegen das SARS-CoV-Spike-Protein und die Nukleokapsidproteine SARS-CoV-2 erkennen (Abbildung 3, Tafeln B, C). Das Nukleokapsidprotein, das innerhalb der Gruppe 2B hoch konserviert ist, weist eine >90%ige Aminosäureidentität zwischen SARS-CoV und SARS-CoV-2 auf. In Übereinstimmung mit den Replikationsergebnissen (Abbildung 3, Tafel A) zeigte SARS-CoV-2 in beiden Vero-Zelltypen ein robustes Nukleokapsidprotein, weniger Protein in HUH7.0- und 293T-Zellen und minimales Protein in A549- und EFK3B-Zellen (Abbildung 3, Tafel B). Der SARS-CoV-Spike-Protein-Antikörper erkannte auch das SARS-CoV-2-Spike-Protein, was auf eine Kreuzreaktivität hinweist (Abbildung 3, Tafel C). In Übereinstimmung mit SARS-CoV wurden mehrere gespaltene und ungespaltene Formen des SARS-CoV-2-Spike-Proteins beobachtet. Das Spaltungsmuster der SARS-Spike-Positivkontrolle aus Calu3-Zellen, einer respiratorischen Zelllinie, variiert geringfügig und könnte auf Unterschiede bei der proteolytischen Spaltung der Spike-Proteine zwischen den beiden Viren hinweisen, da eine Furin-Spaltstelle in SARS-CoV-2 vermutet wird (16). Unterschiede im Zelltyp und in den Bedingungen erschweren jedoch diese Interpretation und weisen auf die Notwendigkeit weiterer Untersuchungen in gleichwertigen Systemen hin. Insgesamt rekapitulieren die Proteinexpressionsdaten von SARS-CoV-Nukleokapsid- und Spike-Protein-Antikörpern die Replikationsergebnisse und weisen darauf hin, dass SARS-CoV-Reagenzien zur Charakterisierung einer SARS-CoV-2-Infektion verwendet werden können.
Abbildung 4
Abbildung 4. Mehrstufige Wachstumskurve für Coronavirus 2 des schweren akuten respiratorischen Syndroms bei einem Patienten mit Coronavirus-Erkrankung, Vereinigte Staaten, 2020. Vero CCL81 (schwarz) und HUH7.0 Zellen (grün) wurden mit einer Multiplizität von…
Schließlich haben wir die Replikationskinetik von SARS-CoV-2 in einer mehrstufigen Wachstumskurve bewertet. Dazu infizierten wir Vero CCL-81- und HUH7.0-Zellen mit SARS-CoV-2 bei einer niedrigen Infektionsmultiplikation (0,1) und untersuchten die virale Replikation alle 6 Stunden über 72 Stunden nach der Inokulation, wobei die zellassoziierten und die überstehenden Kompartimente getrennt erfasst wurden (Abbildung 4). Ähnlich wie SARS-CoV replizierte sich SARS-CoV-2 in Vero-Zellen nach einer anfänglichen Eclipse-Phase schnell und erreichte bis 24 Stunden nach der Infektion 105 TCID50/ml und erreichte einen Spitzenwert von >106 TCID50/ml. Wir beobachteten ähnliche Titer in zellassoziierten und Überstandskompartimenten, was auf einen effizienten Austritt hinweist. Obwohl die Virustiter 48 Stunden nach der Inokulation ihren Höchststand erreichten, wurde der CPE erst 60 Stunden nach der Inokulation beobachtet und erreichte seinen Höhepunkt 72 Stunden nach der Inokulation, was darauf hindeutet, dass infizierte Monolayer geerntet werden sollten, bevor der CPE seinen Höhepunkt erreicht. Die Replikation in HUH7.0-Zellen nahm nach einer anfänglichen Eclipse-Phase ebenfalls rasch zu, erreichte jedoch 24 Stunden nach der Inokulation im intrazellulären Kompartiment mit 2 × 103 TCID50/ml ein Plateau und nahm nach 66 Stunden nach der Inokulation ab. Das Virus wurde im Überstand infizierter HUH7-Zellen bis 36 Stunden nach der Inokulation nicht nachgewiesen und wies zu allen Zeitpunkten niedrigere Titer auf (Abbildung 4). Ein größerer CPE wurde in HUH7.0-Zellen nie beobachtet. Diese Ergebnisse stimmen mit früheren Berichten über SARS-CoV und MERS-CoV überein, die auf eine ähnliche Replikationsdynamik zwischen den zoonotischen CoV-Stämmen schließen lassen (23,24).
