Results
Figure 1
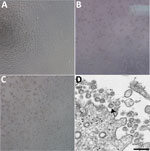
Figure 1. Cytopatisk virkning forårsaget af alvorligt akut respiratorisk syndrom coronavirus 2 fra patient med coronavirus-sygdom, USA, 2020. A-C) Fase-kontrastmikroskopi af Vero-cellemonolag 3 dage efter inokulation: A) Mock,…
En patient blev identificeret med bekræftet COVID-19 i staten Washington den 22. januar 2020. Der blev ikke observeret CPE i mock-inficerede celler (figur 1, panel A). Cykletærskelværdierne (Ct) var 18-20 for NP-prøver og 21-22 for OP-prøver (1). De positive kliniske prøver blev aliquoteret og genfrosset og inokuleret i cellekultur den 22. januar 2020. Vi observerede CPE 2 dage efter inokulation og høstede viralt lysat på dag 3 efter inokulation (figur 1, panel B og C). Vi brugte 50 μL af passage 1 virale lysater til ekstraktion af nukleinsyre for at bekræfte tilstedeværelsen af SARS-CoV-2 ved hjælp af CDC’s molekylærdiagnostiske assay (1). Ct-værdierne for 3 nukleinsyreekstraktioner var 16,0-17,1 for nukleokapsiddel 1, 15,9-17,1 for nukleokapsiddel 2 og 16,2-17,3 for nukleokapsiddel 3, hvilket bekræftede isolering af SARS-CoV-2 (Ct <40 anses for et positivt resultat). Vi testede også ekstrakter for yderligere 33 forskellige respiratoriske patogener ved hjælp af Fast Track 33-assayet. Der blev ikke påvist andre patogener. Identiteten blev desuden understøttet af elektronmikroskopi i tyndt snit (figur 1, panel D). Vi observerede en morfologi og morfogenese, der er karakteristisk for coronavirusser.
Vi anvendte isolater fra den første passage af en OP- og en NP-prøve til helgenomsekventering. Genomerne fra NP-eksemplaret (GenBank accession MT020880) og OP-eksemplaret (GenBank accession nr. MT020881) viste 100 % identitet med hinanden. Isolaterne viste også 100 % identitet med den tilsvarende kliniske prøve (GenBank accession no. MN985325).
Efter den anden passage dyrkede vi ikke OP- og NP-prøverne separat. Vi passerede virusisolatet yderligere 2 gange i Vero CCL-81-celler og titrerede ved at bestemme den 50 % vævskulturinfektiøse dosis (TCID50). Titrene var 8,65 × 106 TCID50/mL for tredje passage og 7,65 × 106 TCID50/mL for fjerde passage.
Vi passerede dette virus i fravær af trypsin. Spike-proteinsekvensen af SARS-CoV-2 har en RRAR-indsættelse ved S1-S2-grænsefladen, som kan blive spaltet af furin (16). Højpatogene aviære influenzavirus har meget basiske furin-spaltningssteder ved grænsefladen mellem hemagglutinininprotein HA1-HA2, som muliggør intracellulær modning af virioner og mere effektiv viral replikation (17). RRAR-indsættelsen i SARS-CoV-2 har muligvis en lignende funktion.
Figur 2
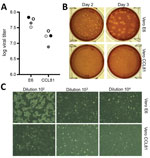
Figur 2. Viral udbredelse og kvantificering af alvorligt akut respiratorisk syndrom coronavirus 2 fra patient med coronavirus-sygdom, USA, 2020. A) To virusbestande med passage 4 (sorte og grå cirkler) blev kvantificeret…
Vi genererede efterfølgende en bestand med fjerde passage af SARS-CoV-2 på VeroE6-celler, en anden føtal rhesusabes nyrecellelinje. Vi sekventerede viralt RNA fra SARS-CoV-2 passage 4-stammen og bekræftede, at det ikke havde nogen nukleotidmutationer sammenlignet med den oprindelige referencesekvens (GenBank accession nr. MN985325). SARS-CoV har vist sig at vokse godt på VeroE6-celler og MERS-CoV på Vero CCL81-celler (18,19). For at etablere et plakatforsøg og bestemme den foretrukne Vero-celletype til kvantificering, titerede vi vores passage 4-stamme på VeroE6- og VeroCCL81-celler. Efter infektion med en fortyndingsserie replikerede SARS-CoV-2 i begge Vero-celletyper; de virale titre var dog en smule højere i VeroE6-celler end i Vero CCL81-celler (figur 2, panel A). Desuden var plakkerne mere tydelige og synlige på Vero E6-celler (figur 2, panel B). Allerede 2 dage efter inokulering producerede VeroE6-cellerne tydelige plaques, der var synlige ved farvning med neutral rødt. I modsætning hertil producerede Vero CCL81-celler mindre tydelige plaques, som lettest kunne kvantificeres ved farvning med neutral rødt 3 dage efter inokulation. På de enkelte plaque-monolag producerede SARS-CoV-2-infektion af Vero E6-celler CPE med områder med celleudrensning (figur 2, panel C). I modsætning hertil havde Vero CCL81-celler områder med døde celler, der var smeltet sammen til plaques, men cellerne blev ikke fjernet. Tilsammen tyder disse resultater på, at VeroE6-celler måske er det bedste valg til forstærkning og kvantificering, men begge Vero-celletyper understøtter forstærkning og replikation af SARS-CoV-2.
Figur 3
Figur 3. Cellelinjer fra patient med coronavirus-sygdom, USA, 2020, modtagelige for SARS-coronavirus 2 (SARS-CoV-2). Cellelinjerne blev inficeret med en høj infektionsmultiplicitet (>5), vasket efter adsorption og…
Da der er indledt forskning med henblik på at undersøge og reagere på SARS-CoV-2, er der behov for oplysninger om cellelinjer og typer, der er modtagelige for infektion. Derfor undersøgte vi SARS-CoV-2’s evne til at inficere og replikere i flere almindelige primat- og menneskecellelinjer, herunder humane adenocarcinomceller (A549), humane leverceller (HUH7.0) og humane embryonale nyreceller (HEK-293T), foruden Vero E6- og Vero CCL81-celler. Vi undersøgte også en tilgængelig stor brun flagermusnyrecellelinje (EFK3B) for SARS-CoV-2 replikationskapacitet. Hver cellelinje blev inokuleret ved høj infektionsmultiplicitet og undersøgt 24 timer efter infektionen (figur 3, panel A). Der blev ikke observeret nogen CPE i nogen af cellelinjerne undtagen i Vero-cellerne, som voksede til >107 PFU 24 timer efter infektionen. I modsætning hertil viste HUH7.0- og 293T-celler kun en beskeden viral replikation, og A549-celler var uforenelige med SARS-CoV-2-infektion. Disse resultater er i overensstemmelse med tidligere fund af modtagelighed for SARS-CoV og tyder på, at andre almindelige dyrkningssystemer, herunder MDCK, HeLa, HEP-2, MRC-5-celler og embryonerede æg, sandsynligvis ikke understøtter replikation af SARS-CoV-2 (20-22). Desuden replicerede SARS-CoV-2 sig ikke i EFK3B-celler fra flagermus, som er modtagelige for MERS-CoV. Tilsammen tyder resultaterne på, at SARS-CoV-2 opretholder en lignende profil som SARS-CoV med hensyn til modtagelige cellelinjer.
Når vi havde etableret robust infektion med SARS-CoV-2 i flere celletyper, evaluerede vi dernæst krydsreaktiviteten af SARS-CoV-antistoffer mod SARS-CoV-2. Celle lysater fra inficerede cellelinjer blev undersøgt med henblik på proteinanalyse; vi fandt, at polyklonalt serum mod SARS-CoV spike-protein og nukleokapsidproteiner genkender SARS-CoV-2 (figur 3, panel B og C). Nukleokapsidproteinet, som er meget konserveret i hele gruppe 2B-familien, har >90% aminosyreidentitet mellem SARS-CoV og SARS-CoV-2. I overensstemmelse med replikationsresultaterne (figur 3, panel A) viste SARS-CoV-2 et robust nukleokapsidprotein i begge Vero-celletyper, mindre protein i HUH7.0- og 293T-celler og et minimalt protein i A549- og EFK3B-celler (figur 3, panel B). Antistoffet til SARS-CoV-spikeprotein genkendte også SARS-CoV-2-spikeprotein, hvilket indikerer krydsreaktivitet (figur 3, panel C). I overensstemmelse med SARS CoV blev der observeret flere spaltede og ikke-spaltede former af SARS-CoV-2 spikeprotein. Spaltningsmønstret for SARS spike-positivkontrollen fra Calu3-celler, en respiratorisk cellelinje, varierer en smule og kunne indikere forskelle mellem den proteolytiske spaltning af spike-proteinerne mellem de to vira på grund af en forudsagt indsættelse af et furin-spaltningssted i SARS-CoV-2 (16). Forskelle i celletype og betingelser komplicerer imidlertid denne fortolkning og viser, at der er behov for yderligere undersøgelser i tilsvarende systemer. Samlet set rekapitulerer proteinekspressionsdataene fra SARS-CoV nukleokapsid- og spikeproteinantistoffer replikationsresultater og indikerer, at SARS-CoV-reagenser kan anvendes til at karakterisere SARS-CoV-2-infektion.
Figur 4
Figur 4. Flertrinsvækstkurve for alvorligt akut respiratorisk syndrom coronavirus 2 fra patient med coronavirussygdom, USA, 2020. Vero CCL81 (sort) og HUH7.0-celler (grøn) blev inficeret ved en multiplicitet på…
Endeligt evaluerede vi replikationskinetikken for SARS-CoV-2 i en flertrinsvækstkurve. Kort fortalt inficerede vi Vero CCL-81- og HUH7.0-celler med SARS-CoV-2 ved en lav infektionsmultiplicitet (0,1) og evaluerede viral replikation hver 6. time i 72 timer efter inokulationen med separate høstninger i de celleassocierede og supernatantkompartmenter (figur 4). I lighed med SARS-CoV replikerede SARS-CoV-2 sig hurtigt i Vero-celler efter en indledende formørkelsesfase og nåede op på 105 TCID50/mL 24 timer efter infektionen og toppede ved >106 TCID50/mL. Vi observerede lignende titre i celleassocierede og supernatantkompartmenter, hvilket indikerede en effektiv udvanding. På trods af maksimale virale titre ved 48 h postinokulering blev der ikke observeret større CPE før 60 h postinokulering og toppede ved 72 h postinokulering, hvilket indikerer, at inficerede monolag bør høstes, før der observeres maksimal CPE. Replikation i HUH7.0-celler øgedes også hurtigt efter en indledende formørkelsesfase, men nåede et plateau efter 24 timer postinokulering i det intracellulære rum ved 2 × 103 TCID50/mL og faldt efter 66 timer postinokulering. Virus blev ikke påvist i supernatanten af inficerede HUH7-celler før 36 timer efter inokulering og udviste lavere titre på alle tidspunkter (figur 4). Der blev aldrig observeret større CPE i HUH7.0-celler. Disse resultater er i overensstemmelse med tidligere rapporter om SARS-CoV og MERS-CoV, som tyder på en lignende replikationsdynamik mellem de zoonotiske CoV-stammer (23,24).
